Más sobre forecasting en: cienciadedatos.net
- Forecasting series temporales con machine learning
- Modelos ARIMA y SARIMAX
- Forecasting series temporales con gradient boosting: XGBoost, LightGBM y CatBoost
- Global Forecasting: Multi-series forecasting
- Forecasting de la demanda eléctrica con machine learning
- Forecasting con deep learning
- Forecasting de visitas a página web con machine learning
- Forecasting del precio de Bitcoin
- Forecasting probabilístico
- Forecasting de demanda intermitente
- Reducir el impacto del Covid en modelos de forecasting
- Modelar series temporales con tendencia utilizando modelos de árboles

Introducción¶
Al tratar de anticipar valores futuros de una serie temporal, la mayoría de los modelos de forecasting intentan predecir cuál será el valor más probable, esto se llama point-forecasting. Aunque conocer de antemano el valor esperado de una serie temporal es útil en casi todos los casos de negocio, este tipo de predicción no proporciona información sobre la confianza del modelo ni sobre la incertidumbre de sus predicciones.
El forecasting probabilístico, a diferencia del point-forecasting, es una familia de técnicas que permiten predecir la distribución esperada de la variable respuesta en lugar de un único valor puntual. Este tipo de forecasting proporciona información muy valiosa ya que permite crear intervalos de predicción, es decir, el rango de valores donde es más probable que pueda estar el valor real. Más formalmente, un intervalo de predicción define el intervalo dentro del cual se espera encontrar el verdadero valor de la variable respuesta con una determinada probabilidad.
Skforecast implementa varios métodos para la predicción probabilística:
Bootstrapped residuals: El bootstrapping es una técnica estadística que permite estimar la distribución de un estadístico al muestrear repetidamente los datos con reemplazo. En el contexto del forecasting, aplicar bootstrapping a los residuos de un modelo permite estimar la distribución de los errores, lo que facilita la construcción de intervalos de predicción.
Conformal prediction: conformal prediction engloba un conjunto de técnicas que permiten generar intervalos de predicción garantizando una determinada cobertura. Se basa en combinar las predicciones puntuales de un modelo de forecasting con sus residuales históricos (diferencias entre predicciones previas y valores observados). Estos residuales permiten estimar la incertidumbre en la predicción y ajustar la amplitud del intervalo entorno a la predicción puntual. Skforecast utiliza Split Conformal Prediction (SCP).
Además, los métodos conformal pueden calibrar intervalos de predicción generados por otras técnicas, como la regresión cuantílica o bootstrapping. En estos casos, el método conformal ajusta los intervalos para garantizar que sigan siendo válidos con respecto a una determinada de cobertura.
Regresión cuantílica (Quantile regression): La regresión cuantílica permite modelar los cuantiles de una variable de respuesta. Al combinar las predicciones de dos modelos de regresión cuantílica, se puede construir un intervalo de predicción en el que cada modelo estima uno de los límites. Por ejemplo, entrenar modelos con $Q = 0.1$ y $Q = 0.9$ produce un intervalo de predicción del 80% ($90\% - 10\% = 80\%$).
⚠️ Warning
Tal y como describe Rob J Hyndman en su blog, en los casos reales, casi todos los intervalos de predicción resultan ser demasiado estrechos. Por ejemplo, intervalos teóricos del 95% solo suelen conseguir una cobertura real de entre el 71% y el 87%. Este fenómeno surge debido a que estos intervalos no contemplan todas las fuentes de incertidumbre que, en el caso de modelos de *forecasting*, suelen ser de 4 tipos: el término de error aleatorio, las estimación de parámetros, la elección del modelo y el proceso de predicción de valores futuros. Cuando se calculan intervalos de predicción para modelos de series temporales, generalmente solo se tiene en cuenta el primero de ellos. Por lo tanto, es recomendable utilizar datos de test para validar la cobertura real del intervalo y no confiar únicamente en la esperada.
💡 Tip
Este es el primero de una serie de documentos sobre forecasting probabilístico.
Bootstrapped Residuals¶
El error en la predicción del siguiente valor de una serie (one-step-ahead forecast) se define como $e_t = y_t - \hat{y}_{t|t-1}$. Asumiendo que los errores futuros serán similares a los errores pasados, es posible simular diferentes predicciones tomando muestras de los errores vistos previamente en el pasado (es decir, los residuos) y agregándolos a las predicciones.
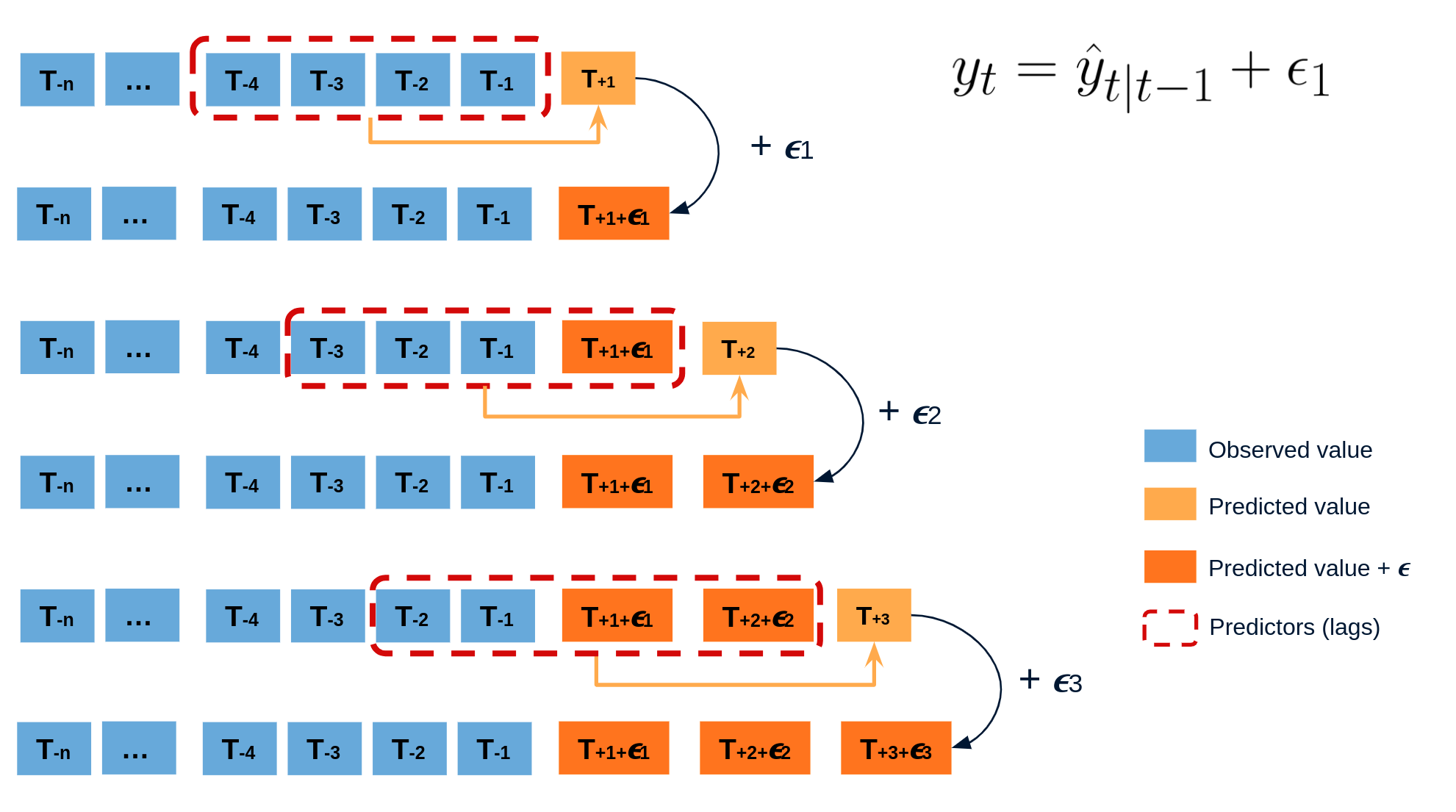
Al hacer esto repetidamente, se crea una colección de predicciones ligeramente diferentes (posibles caminos futuros), que representan la varianza esperada en el proceso de forecasting.
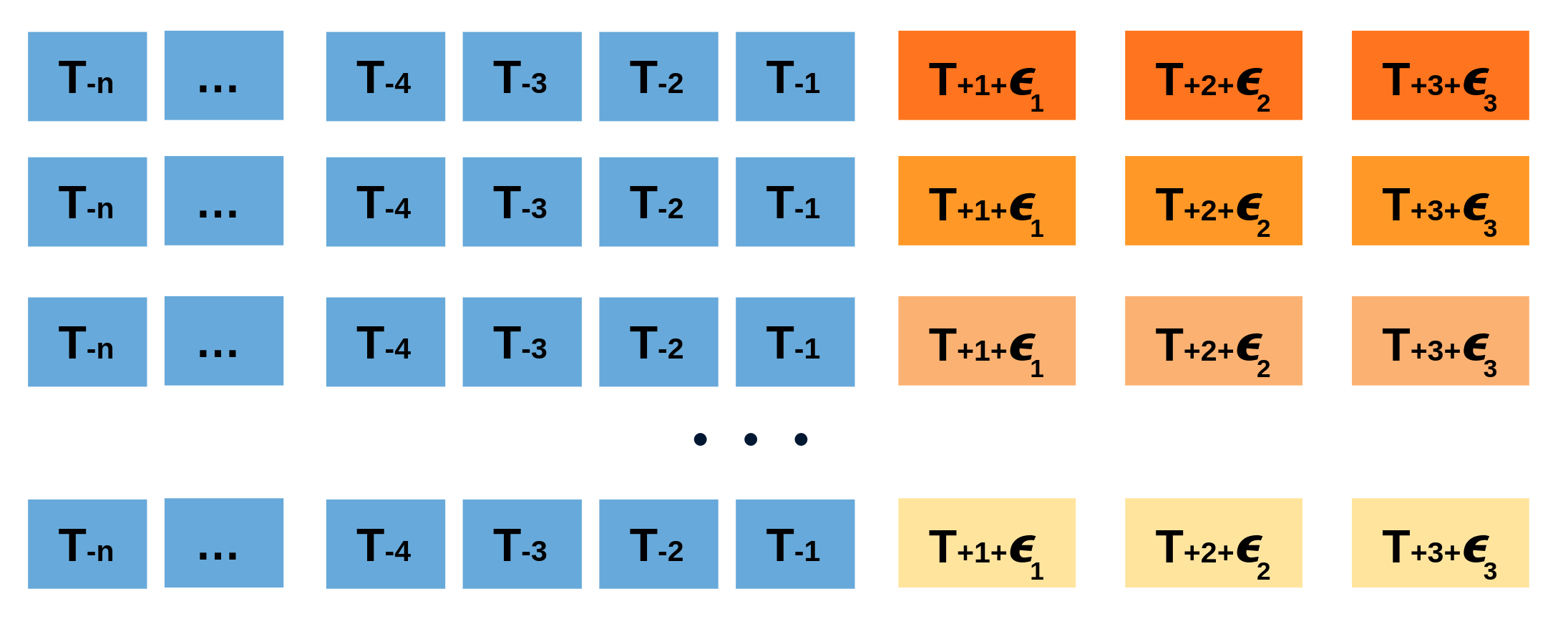
Finalmente, los intervalos de predicción se crean calculando los percentiles $\alpha/2$ y $1−\alpha/2$ de los datos simulados en cada horizonte de predicción.
Animación del proceso de predicción probabilística mediante bootstrapping.
La principal ventaja de esta estrategia es que solo requiere de un único modelo para estimar cualquier intervalo. El inconveniente es la necesidad de ejecutar cientos o miles de iteraciones de bootstrapping lo cual resulta muy costoso desde el punto de vista computacional y no siempre es posible.
Librerías y datos¶
# Preprocesado de datos
# ==============================================================================
import numpy as np
import pandas as pd
from skforecast.datasets import fetch_dataset
# Plots
# ==============================================================================
import matplotlib.pyplot as plt
import plotly.graph_objects as go
import plotly.io as pio
import plotly.offline as poff
pio.templates.default = "seaborn"
pio.renderers.default = 'notebook'
poff.init_notebook_mode(connected=True)
plt.style.use('seaborn-v0_8-darkgrid')
from skforecast.plot import plot_residuals
from pprint import pprint
# Modelado y Forecasting
# ==============================================================================
import skforecast
from lightgbm import LGBMRegressor
from sklearn.pipeline import make_pipeline
from feature_engine.datetime import DatetimeFeatures
from feature_engine.creation import CyclicalFeatures
from skforecast.recursive import ForecasterRecursive
from skforecast.direct import ForecasterDirect
from skforecast.preprocessing import RollingFeatures
from skforecast.model_selection import TimeSeriesFold, backtesting_forecaster, bayesian_search_forecaster
from skforecast.metrics import calculate_coverage, create_mean_pinball_loss
# Configuración
# ==============================================================================
import warnings
warnings.filterwarnings('once')
color = '\033[1m\033[38;5;208m'
print(f"{color}Version skforecast: {skforecast.__version__}")
Version skforecast: 0.20.0
# Descarga de datos
# ==============================================================================
data = fetch_dataset(name='bike_sharing', raw=False)
data = data[['users', 'temp', 'hum', 'windspeed', 'holiday']]
data = data.loc['2011-04-01 00:00:00':'2012-10-20 23:00:00', :].copy()
data.head(3)
╭───────────────────────────────── bike_sharing ──────────────────────────────────╮ │ Description: │ │ Hourly usage of the bike share system in the city of Washington D.C. during the │ │ years 2011 and 2012. In addition to the number of users per hour, information │ │ about weather conditions and holidays is available. │ │ │ │ Source: │ │ Fanaee-T,Hadi. (2013). Bike Sharing Dataset. UCI Machine Learning Repository. │ │ https://doi.org/10.24432/C5W894. │ │ │ │ URL: │ │ https://raw.githubusercontent.com/skforecast/skforecast- │ │ datasets/main/data/bike_sharing_dataset_clean.csv │ │ │ │ Shape: 17544 rows x 11 columns │ ╰─────────────────────────────────────────────────────────────────────────────────╯
| users | temp | hum | windspeed | holiday | |
|---|---|---|---|---|---|
| date_time | |||||
| 2011-04-01 00:00:00 | 6.0 | 10.66 | 100.0 | 11.0014 | 0.0 |
| 2011-04-01 01:00:00 | 4.0 | 10.66 | 100.0 | 11.0014 | 0.0 |
| 2011-04-01 02:00:00 | 7.0 | 10.66 | 93.0 | 12.9980 | 0.0 |
Se añaden variables adicionales creadas a partir del calendario.
# Variables exógenas basadas en el calendario
# ==============================================================================
features_to_extract = ['month', 'week', 'day_of_week', 'hour']
calendar_transformer = DatetimeFeatures(
variables = 'index',
features_to_extract = features_to_extract,
drop_original = False,
)
# Codificación de variables cíclicas
# ==============================================================================
features_to_encode = ['month', 'week', 'day_of_week', 'hour']
max_values = {"month": 12, "week": 52, "day_of_week": 7, "hour": 24}
cyclical_encoder = CyclicalFeatures(
variables = features_to_encode,
max_values = max_values,
drop_original = True
)
exog_transformer = make_pipeline(calendar_transformer, cyclical_encoder)
data = exog_transformer.fit_transform(data)
exog_features = data.columns.difference(['users']).tolist()
data.head(3)
| users | temp | hum | windspeed | holiday | month_sin | month_cos | week_sin | week_cos | day_of_week_sin | day_of_week_cos | hour_sin | hour_cos | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| date_time | |||||||||||||
| 2011-04-01 00:00:00 | 6.0 | 10.66 | 100.0 | 11.0014 | 0.0 | 0.866025 | -0.5 | 1.0 | 6.123234e-17 | -0.433884 | -0.900969 | 0.000000 | 1.000000 |
| 2011-04-01 01:00:00 | 4.0 | 10.66 | 100.0 | 11.0014 | 0.0 | 0.866025 | -0.5 | 1.0 | 6.123234e-17 | -0.433884 | -0.900969 | 0.258819 | 0.965926 |
| 2011-04-01 02:00:00 | 7.0 | 10.66 | 93.0 | 12.9980 | 0.0 | 0.866025 | -0.5 | 1.0 | 6.123234e-17 | -0.433884 | -0.900969 | 0.500000 | 0.866025 |
Para facilitar el entrenamiento de los modelos, la búsqueda de hiperparámetros óptimos y la evaluación de su capacidad predictiva, los datos se dividen en tres conjuntos separados: entrenamiento, validación y prueba.
# Partición de datos en entrenamiento-validación-test
# ==============================================================================
end_train = '2012-06-30 23:59:00'
end_validation = '2012-10-01 23:59:00'
data_train = data.loc[: end_train, :]
data_val = data.loc[end_train:end_validation, :]
data_test = data.loc[end_validation:, :]
print(f"Dates train : {data_train.index.min()} --- {data_train.index.max()} (n={len(data_train)})")
print(f"Dates validacion : {data_val.index.min()} --- {data_val.index.max()} (n={len(data_val)})")
print(f"Dates test : {data_test.index.min()} --- {data_test.index.max()} (n={len(data_test)})")
Dates train : 2011-04-01 00:00:00 --- 2012-06-30 23:00:00 (n=10968) Dates validacion : 2012-07-01 00:00:00 --- 2012-10-01 23:00:00 (n=2232) Dates test : 2012-10-02 00:00:00 --- 2012-10-20 23:00:00 (n=456)
# Gráfico de la serie temporal
# ==============================================================================
fig = go.Figure()
fig.add_trace(go.Scatter(x=data_train.index, y=data_train['users'], mode='lines', name='Train'))
fig.add_trace(go.Scatter(x=data_val.index, y=data_val['users'], mode='lines', name='Validation'))
fig.add_trace(go.Scatter(x=data_test.index, y=data_test['users'], mode='lines', name='Test'))
fig.update_layout(
title = 'Número de usuarios',
xaxis_title="Fecha",
yaxis_title="Usuarios",
width=800,
height=400,
margin=dict(l=20, r=20, t=35, b=20),
legend=dict(orientation="h", yanchor="top", y=1, xanchor="left", x=0.001),
)
fig.show()
Intervalos con residuos in-sample¶
Los intervalos se pueden calcular utilizando los residuos in-sample (residuos del conjunto de entrenamiento), ya sea llamando al método predict_interval(), o realizando un procedimiento completo de backtesting. Sin embargo, esto puede resultar en intervalos que son demasiado estrechos (demasiado optimistas).
✏️ Note
Los hiperparámetros utilizados en este ejemplo han sido previamente optimizados mediante un proceso de búsqueda bayesiana. Para obtener más información sobre este proceso, consulte Hyperparameter tuning and lags selection.
# Crear y entrenar modelo
# ==============================================================================
params = {
"max_depth": 7,
"n_estimators": 300,
"learning_rate": 0.06,
"verbose": -1,
"random_state": 15926
}
lags = [1, 2, 3, 23, 24, 25, 167, 168, 169]
window_features = RollingFeatures(stats=["mean"], window_sizes=24 * 3)
forecaster = ForecasterRecursive(
estimator = LGBMRegressor(**params),
lags = lags,
window_features = window_features,
)
forecaster.fit(
y = data.loc[:end_validation, 'users'],
exog = data.loc[:end_validation, exog_features],
store_in_sample_residuals = True
)
# In-sample residuals stored during fit
# ==============================================================================
print("Amount of residuals stored:", len(forecaster.in_sample_residuals_))
forecaster.in_sample_residuals_
Amount of residuals stored: 10000
array([ 42.02327339, -4.75342728, -39.26777553, ..., -3.54886809,
-41.20842177, -13.42207696], shape=(10000,))
Se utiliza la función backtesting_forecaster() para generar los intervalos de predicción de todo el conjunto de test. Los principales argumentos de esta función son:
use_in_sample_residuals: cuando esTrue, los residuos in-sample se utilizan para calcular los intervalos de predicción. Dado que estos residuos se obtienen del conjunto de entrenamiento, siempre están disponibles, pero suelen dar lugar a intervalos demasiado optimistas. Cuando esFalse, los residuos out-sample se utilizan para calcular los intervalos de predicción. Estos residuos se obtienen del conjunto de validación y solo están disponibles si se ha llamado al métodoset_out_sample_residuals(). Se recomienda utilizar los residuos out-sample para lograr la cobertura deseada.interval: la cobertura deseada de los intervalos de predicción. Por ejemplo,[10, 90]significa que los intervalos de predicción se calculan para los percentiles 10 y 90, lo que da como resultado una cobertura teórica del 80%.interval_method:el método utilizado para calcular los intervalos de predicción. Las opciones disponibles sonbootstrappingyconformal.n_boot: el número de muestras bootstrap que se utilizan para estimar los intervalos de predicción cuandointerval_method = 'bootstrapping'. Cuanto mayor sea el número de muestras, más precisos serán los intervalos de predicción, pero mayor el tiempo necesario.
# Backtesting con intervalos en los datos de test usando residuos in-sample
# ==============================================================================
cv = TimeSeriesFold(steps = 24, initial_train_size = len(data.loc[:end_validation]))
metric, predictions = backtesting_forecaster(
forecaster = forecaster,
y = data['users'],
exog = data[exog_features],
cv = cv,
metric = 'mean_absolute_error',
interval = [10, 90], # 80% prediction interval
interval_method = 'bootstrapping',
n_boot = 150,
use_in_sample_residuals = True, # Use in-sample residuals
use_binned_residuals = False,
)
predictions.head(5)
0%| | 0/19 [00:00<?, ?it/s]
| fold | pred | lower_bound | upper_bound | |
|---|---|---|---|---|
| 2012-10-02 00:00:00 | 0 | 58.387527 | 32.799491 | 78.248425 |
| 2012-10-02 01:00:00 | 0 | 17.870302 | -7.253084 | 50.190172 |
| 2012-10-02 02:00:00 | 0 | 7.901576 | -17.057776 | 37.596425 |
| 2012-10-02 03:00:00 | 0 | 5.414332 | -16.809510 | 40.194483 |
| 2012-10-02 04:00:00 | 0 | 10.379630 | -12.696466 | 56.904041 |
# Function to plot predicted intervals
# ======================================================================================
def plot_predicted_intervals(
predictions: pd.DataFrame,
y_true: pd.DataFrame,
target_variable: str,
initial_x_zoom: list = None,
title: str = None,
xaxis_title: str = None,
yaxis_title: str = None,
):
"""
Plot predicted intervals vs real values
Parameters
----------
predictions : pandas DataFrame
Predicted values and intervals.
y_true : pandas DataFrame
Real values of target variable.
target_variable : str
Name of target variable.
initial_x_zoom : list, default `None`
Initial zoom of x-axis, by default None.
title : str, default `None`
Title of the plot, by default None.
xaxis_title : str, default `None`
Title of x-axis, by default None.
yaxis_title : str, default `None`
Title of y-axis, by default None.
"""
fig = go.Figure([
go.Scatter(name='Prediction', x=predictions.index, y=predictions['pred'], mode='lines'),
go.Scatter(name='Real value', x=y_true.index, y=y_true[target_variable], mode='lines'),
go.Scatter(
name='Upper Bound', x=predictions.index, y=predictions['upper_bound'],
mode='lines', marker=dict(color="#444"), line=dict(width=0), showlegend=False
),
go.Scatter(
name='Lower Bound', x=predictions.index, y=predictions['lower_bound'],
marker=dict(color="#444"), line=dict(width=0), mode='lines',
fillcolor='rgba(68, 68, 68, 0.3)', fill='tonexty', showlegend=False
)
])
fig.update_layout(
title=title, xaxis_title=xaxis_title, yaxis_title=yaxis_title, width=800,
height=400, margin=dict(l=20, r=20, t=35, b=20), hovermode="x",
xaxis=dict(range=initial_x_zoom),
legend=dict(orientation="h", yanchor="top", y=1.1, xanchor="left", x=0.001)
)
fig.show()
# Gráfico intervalos
# ==============================================================================
plot_predicted_intervals(
predictions = predictions,
y_true = data_test,
target_variable = "users",
xaxis_title = "Date time",
yaxis_title = "users",
)
# Cobertura del intervalo en los datos de test
# ==============================================================================
coverage = calculate_coverage(
y_true = data.loc[end_validation:, 'users'],
lower_bound = predictions["lower_bound"],
upper_bound = predictions["upper_bound"]
)
print(f"Cobertura del intervalo: {round(100 * coverage, 2)} %")
# Area del intervalo
# ==============================================================================
area = (predictions["upper_bound"] - predictions["lower_bound"]).sum()
print(f"Area of the interval: {round(area, 2)}")
Cobertura del intervalo: 61.84 % Area of the interval: 43177.89
Los intervalos de predicción presentan un exceso de confianza, ya que tienden a ser demasiado estrechos, lo que da lugar a una cobertura real inferior a la cobertura teórica (80%). Esto se debe a la tendencia de los residuos de entrenamiento (in-sample) a sobrestimar la capacidad de predicción del modelo.
# Almacenar las predicciones para su posterior uso
# ==============================================================================
predictions_in_sample_residuals = predictions.copy()
Residuos Out-sample (no condicionados a los valores predichos)¶
Para evitar el problema de los intervalos demasiado optimistas, es posible utilizar los residuos out-sample (residuos de un conjunto de validación no vistos durante el entrenamiento). Estos residuos se pueden obtener a través de un proceso de backtesting.
# Backtesting con datos de validación para obtener residuos out-sample
# ==============================================================================
cv = TimeSeriesFold(steps = 24, initial_train_size = len(data.loc[:end_train]))
_, predictions_val = backtesting_forecaster(
forecaster = forecaster,
y = data.loc[:end_validation, 'users'],
exog = data.loc[:end_validation, exog_features],
cv = cv,
metric = 'mean_absolute_error',
)
0%| | 0/93 [00:00<?, ?it/s]
# Distribución residuos out-sample
# ==============================================================================
residuals = data.loc[predictions_val.index, 'users'] - predictions_val['pred']
print(pd.Series(np.where(residuals < 0, 'negative', 'positive')).value_counts())
plt.rcParams.update({'font.size': 8})
_ = plot_residuals(residuals=residuals, figsize=(7, 4))
positive 1297 negative 935 Name: count, dtype: int64
Con el método set_out_sample_residuals(), los residuos out-sample se almacenan en el objeto forecaster para que puedan ser utilizados para calibrar los intervalos de predicción.
# Almacenar residuos out-sample en el forecaster
# ==============================================================================
forecaster.set_out_sample_residuals(
y_true = data.loc[predictions_val.index, 'users'],
y_pred = predictions_val['pred']
)
Ahora que los nuevos residuos se han añadido al objeto forecaster, los intervalos de predicción se pueden calcular utilizando use_in_sample_residuals = False.
# Backtesting con intervalos en los datos de test usando residuos out-sample
# ==============================================================================
cv = TimeSeriesFold(steps = 24, initial_train_size = len(data.loc[:end_validation]))
metric, predictions = backtesting_forecaster(
forecaster = forecaster,
y = data['users'],
exog = data[exog_features],
cv = cv,
metric = 'mean_absolute_error',
interval = [10, 90], # 80% prediction interval
interval_method = 'bootstrapping',
n_boot = 150,
use_in_sample_residuals = False, # Use out-sample residuals
use_binned_residuals = False,
)
predictions.head(3)
0%| | 0/19 [00:00<?, ?it/s]
| fold | pred | lower_bound | upper_bound | |
|---|---|---|---|---|
| 2012-10-02 00:00:00 | 0 | 58.387527 | 30.961801 | 133.296285 |
| 2012-10-02 01:00:00 | 0 | 17.870302 | -14.031592 | 132.635745 |
| 2012-10-02 02:00:00 | 0 | 7.901576 | -32.265023 | 142.360525 |
# Gráfico intervalos
# ==============================================================================
plot_predicted_intervals(
predictions = predictions,
y_true = data_test,
target_variable = "users",
xaxis_title = "Date time",
yaxis_title = "users",
)
# Cobertura del intervalo en los datos de test
# ==============================================================================
coverage = calculate_coverage(
y_true = data.loc[end_validation:, 'users'],
lower_bound = predictions["lower_bound"],
upper_bound = predictions["upper_bound"]
)
print(f"Cobertura del intervalo: {round(100*coverage, 2)} %")
# Area del intervalo
# ==============================================================================
area = (predictions["upper_bound"] - predictions["lower_bound"]).sum()
print(f"Area del intervalo: {round(area, 2)}")
Cobertura del intervalo: 83.33 % Area del intervalo: 101257.22
Los intervalos de predicción obtenidos utilizando los residuos out-sample son considerablemente más amplios que los basados en los residuos in-sample, lo que da como resultado una cobertura empírica más cercana a la cobertura nominal. Sin embargo, al examinar el gráfico, es fácil ver que los intervalos son especialmente amplios cuando los valores predichos son bajos, lo que indica que el modelo no es capaz de localizar correctamente la incertidumbre de sus predicciones.
# Almacenar las predicciones para su posterior uso
# ==============================================================================
predictions_out_sample_residuals = predictions.copy()
Intervalos condicionados a los valores predichos (binned residuals)¶
El proceso de bootstrapping asume que los residuos se distribuyen de forma independiente por lo que pueden utilizarse sin tener en cuenta del valor predicho. En realidad, esto rara vez es cierto; en la mayoría de los casos, la magnitud de los residuos está correlacionado con la magnitud del valor predicho. En este caso, por ejemplo, difícilmente cabría esperar que el error fuera el mismo cuando el número previsto de usuarios es cercano a cero que cuando es de varios cientos.
Para tener en cuenta esta dependencia, skforecast permite distribuir los residuos en K intervalos, en los que cada intervalo está asociado a un rango de valores predichos. Con esta estrategia, el proceso de bootstrapping muestrea los residuos de diferentes intervalos en función del valor predicho, lo que puede mejorar la cobertura del intervalo y ajustar su anchura cuando sea necesario, permitiendo que el modelo distribuya mejor la incertidumbre de sus predicciones.
Para que el forecaster pueda agrupar los residuos out-sample, los valores predichos se pasan al método set_out_sample_residuals() junto con los residuos. Internamente, skforecast utiliza la clase QuantileBinner para agrupar los datos en intervalos basados en cuantiles utilizando numpy.percentile. Esta clase es similar a KBinsDiscretizer pero más rápida para agrupar datos en intervalos basados en cuantiles. Los intervalos se definen siguiendo la convención: bins[i-1] <= x < bins[i]. El proceso de binning puede ajustarse utilizando el argumento binner_kwargs del objeto Forecaster.
# Crear forecaster
# ==============================================================================
forecaster = ForecasterRecursive(
estimator = LGBMRegressor(**params),
lags = lags,
binner_kwargs = {'n_bins': 5}
)
forecaster.fit(
y = data.loc[:end_validation, 'users'],
exog = data.loc[:end_validation, exog_features],
store_in_sample_residuals = True
)
Durante el proceso de entrenamiento, el forecaster utiliza las predicciones in-sample para definir los intervalos en los que se almacenan los residuos en función del valor predicho al que están relacionados (binner_intervals_). Por ejemplo, si el bin "0" tiene un intervalo de (5.5, 10.7), significa que almacenará los residuos de las predicciones que caigan dentro de ese intervalo.
Cuando se calculan los intervalos de predicción, los residuos se muestrean del bin correspondiente al valor predicho. De esta forma, el modelo puede ajustar la anchura de los intervalos en función del valor predicho, lo que puede ayudar a distribuir mejor la incertidumbre de las predicciones.
# Intervalos de los bins
# ==============================================================================
pprint(forecaster.binner_intervals_)
{0: (-0.8943794883271247, 30.676160623625577),
1: (30.676160623625577, 120.49652689276296),
2: (120.49652689276296, 209.69596300954962),
3: (209.69596300954962, 338.3331511270013),
4: (338.3331511270013, 955.802117392104)}
El método set_out_sample_residuals() agrupa los residuos según los intervalos aprendidos durante el entrenamiento. Para evitar utilizar demasiada memoria, el número de residuos almacenados por intervalo se limita a 10_000 // self.binner.n_bins_.
# Almacenar residuos out-sample en el forecaster
# ==============================================================================
forecaster.set_out_sample_residuals(
y_true = data.loc[predictions_val.index, 'users'],
y_pred = predictions_val['pred']
)
# Número de residuos por bin
# ==============================================================================
for k, v in forecaster.out_sample_residuals_by_bin_.items():
print(f" Bin {k}: n={len(v)}")
Bin 0: n=321 Bin 1: n=315 Bin 2: n=310 Bin 3: n=534 Bin 4: n=752
# Distribución de los residuos por bin
# ==============================================================================
out_sample_residuals_by_bin_df = pd.DataFrame(
dict([(k, pd.Series(v)) for k, v in forecaster.out_sample_residuals_by_bin_.items()])
)
fig, ax = plt.subplots(figsize=(6, 3))
out_sample_residuals_by_bin_df.boxplot(ax=ax)
ax.set_title("Distribución de los residuos por bin")
ax.set_xlabel("Bin")
ax.set_ylabel("Resoduals");
El gráfico de cajas muestra cómo la dispersión y magnitud de los residuos difieren en función del valor predicho. Los residuos son mayores y más dispersos cuando el valor predicho es mayor (bin más alto), lo que es consistente con la intuición de que los errores tienden a ser mayores cuando el valor predicho es mayor.
# Summary información de los bins
# ======================================================================================
bins_summary = {
key: pd.DataFrame(value).describe().T.assign(bin=key)
for key, value
in forecaster.out_sample_residuals_by_bin_.items()
}
bins_summary = pd.concat(bins_summary.values()).set_index("bin")
bins_summary.insert(0, "interval", bins_summary.index.map(forecaster.binner_intervals_))
bins_summary['interval'] = bins_summary['interval'].apply(lambda x: np.round(x, 2))
bins_summary
| interval | count | mean | std | min | 25% | 50% | 75% | max | |
|---|---|---|---|---|---|---|---|---|---|
| bin | |||||||||
| 0 | [-0.89, 30.68] | 321.0 | 1.113833 | 12.168815 | -19.168802 | -4.060555 | -0.435406 | 3.392902 | 150.505840 |
| 1 | [30.68, 120.5] | 315.0 | 1.560517 | 31.195637 | -77.399525 | -13.424717 | -3.511859 | 10.819653 | 356.446585 |
| 2 | [120.5, 209.7] | 310.0 | 17.209087 | 51.763411 | -147.240066 | -7.478638 | 15.740146 | 38.113001 | 300.170968 |
| 3 | [209.7, 338.33] | 534.0 | 14.883687 | 69.231176 | -241.322880 | -18.266984 | 10.051186 | 42.654985 | 464.619346 |
| 4 | [338.33, 955.8] | 752.0 | 18.740171 | 105.830048 | -485.278643 | -23.429556 | 25.356507 | 77.634913 | 382.818946 |
Por último, los intervalos de predicción para los datos de test se estiman mediante el proceso de backtesting, con los residuos out-sample condicionados a los valores predichos.
# Backtesting con intervalos en los datos de test usando residuos out-sample
# ==============================================================================
cv = TimeSeriesFold(steps = 24, initial_train_size = len(data.loc[:end_validation]))
metric, predictions = backtesting_forecaster(
forecaster = forecaster,
y = data['users'],
exog = data[exog_features],
cv = cv,
metric = 'mean_absolute_error',
interval = [10, 90], # 80% prediction interval
interval_method = 'bootstrapping',
n_boot = 150,
use_in_sample_residuals = False, # Use out-sample residuals
use_binned_residuals = True, # Residuals conditioned on predicted values
)
predictions.head(3)
0%| | 0/19 [00:00<?, ?it/s]
| fold | pred | lower_bound | upper_bound | |
|---|---|---|---|---|
| 2012-10-02 00:00:00 | 0 | 60.399647 | 38.496874 | 92.876947 |
| 2012-10-02 01:00:00 | 0 | 17.617464 | 9.065502 | 43.844047 |
| 2012-10-02 02:00:00 | 0 | 9.002365 | -0.636921 | 26.285576 |
# Gráfico intervalos
# ==============================================================================
plot_predicted_intervals(
predictions = predictions,
y_true = data_test,
target_variable = "users",
xaxis_title = "Date time",
yaxis_title = "users",
)
# Cobertura del intervalo en los datos de test
# ==============================================================================
coverage = calculate_coverage(
y_true = data.loc[end_validation:, 'users'],
lower_bound = predictions["lower_bound"],
upper_bound = predictions["upper_bound"]
)
print(f"Cobertura del intervalo: {round(100*coverage, 2)} %")
# Area del intervalo
# ==============================================================================
area = (predictions["upper_bound"] - predictions["lower_bound"]).sum()
print(f"Area del intervalo: {round(area, 2)}")
Cobertura del intervalo: 86.4 % Area del intervalo: 95072.24
Cuando se utilizan residuos out-sample condicionados al valor predicho, el área del intervalo se reduce significativamente y la incertidumbre se asigna principalmente a las predicciones con valores altos. Sin embargo, la cobertura empírica sigue siendo superior a la cobertura esperada, lo que significa que los intervalos estimados son conservadores.
El siguiente gráfico compara los intervalos de predicción obtenidos utilizando los residuos in-sample, out-sample y out-sample condicionados a los valores predichos.
# Intervalos utilizando: in-sample residuals, out-sample residuals and binned residuals
# ==============================================================================
predictions_out_sample_residuals_binned = predictions.copy()
fig, ax = plt.subplots(figsize=(8, 4))
ax.fill_between(
data_test.index,
predictions_out_sample_residuals["lower_bound"],
predictions_out_sample_residuals["upper_bound"],
color='gray',
alpha=0.9,
label='Out-sample residuals',
zorder=1
)
ax.fill_between(
data_test.index,
predictions_out_sample_residuals_binned["lower_bound"],
predictions_out_sample_residuals_binned["upper_bound"],
color='#fc4f30',
alpha=0.7,
label='Out-sample binned residuals',
zorder=2
)
ax.fill_between(
data_test.index,
predictions_in_sample_residuals["lower_bound"],
predictions_in_sample_residuals["upper_bound"],
color='#30a2da',
alpha=0.9,
label='In-sample residuals',
zorder=3
)
ax.set_xlim(pd.to_datetime(["2012-10-08 00:00:00", "2012-10-15 00:00:00"]))
ax.set_title("Prediction intervals with different residuals", fontsize=12)
ax.legend();
⚠️ Warning
Forecasting probabilístico en producción
La correcta estimación de los intervalos de predicción depende de que los residuos sean representativos de los errores futuros. Por esta razón, se deben utilizar los residuos *out-of-sample*. Sin embargo, la dinámica de las series y los modelos pueden cambiar con el tiempo, por lo que es importante monitorizar y actualizar regularmente los residuos. Esto se puede hacer fácilmente utilizando el método set_out_sample_residuals().
Predicción de múltiples intervalos¶
La función backtesting_forecaster no solo permite estimar un único intervalo, sino también estimar múltiples cuantiles (percentiles) a partir de los cuales se pueden construir múltiples intervalos de predicción. Esto es útil para evaluar la calidad de los intervalos de predicción para un rango de probabilidades. Además, tiene casi ningún costo computacional adicional en comparación con la estimación de un solo intervalo.
A continuación, se predicen varios percentiles y a partir de estos, se crean intervalos de predicción para diferentes niveles de cobertura nominal - 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% y 95% - y se evalúa su cobertura real.
# Intervalos de predicción para varios porcentajes de cobertura
# ==============================================================================
quantiles = [2.5, 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, 90, 95, 97.5]
intervals = [[2.5, 97.5], [5, 95], [10, 90], [15, 85], [20, 80], [30, 70], [35, 65], [40, 60], [45, 55]]
observed_coverages = []
observed_areas = []
metric, predictions = backtesting_forecaster(
forecaster = forecaster,
y = data['users'],
exog = data[exog_features],
cv = cv,
metric = 'mean_absolute_error',
interval = quantiles,
interval_method = 'bootstrapping',
n_boot = 150,
use_in_sample_residuals = False, # Use out-sample residuals
use_binned_residuals = True
)
predictions.head()
0%| | 0/19 [00:00<?, ?it/s]
| fold | pred | p_2.5 | p_5 | p_10 | p_15 | p_20 | p_25 | p_30 | p_35 | ... | p_55 | p_60 | p_65 | p_70 | p_75 | p_80 | p_85 | p_90 | p_95 | p_97.5 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 2012-10-02 00:00:00 | 0 | 60.399647 | 26.540465 | 32.237144 | 38.496874 | 42.515102 | 46.248204 | 48.730080 | 49.747541 | 50.830767 | ... | 59.877370 | 61.388407 | 63.572870 | 66.179578 | 70.417796 | 77.232456 | 82.866190 | 92.876947 | 109.483516 | 118.113906 |
| 2012-10-02 01:00:00 | 0 | 17.617464 | 4.028027 | 6.993912 | 9.065502 | 10.218569 | 11.732744 | 13.290492 | 15.065701 | 15.542016 | ... | 19.700862 | 21.330240 | 22.453599 | 25.298972 | 28.853490 | 32.110319 | 35.993433 | 43.844047 | 68.261638 | 77.072027 |
| 2012-10-02 02:00:00 | 0 | 9.002365 | -5.454406 | -2.921308 | -0.636921 | 2.627889 | 4.072849 | 5.556213 | 6.372106 | 7.170950 | ... | 11.673119 | 12.670580 | 14.060076 | 15.750417 | 17.660921 | 19.929581 | 23.416450 | 26.285576 | 41.378533 | 52.759679 |
| 2012-10-02 03:00:00 | 0 | 5.306644 | -7.830956 | -6.205384 | -1.102749 | -0.500265 | 1.101968 | 2.283863 | 3.161327 | 4.006352 | ... | 6.650199 | 7.561984 | 8.280507 | 9.689953 | 10.880484 | 11.786155 | 14.400937 | 17.854639 | 24.489084 | 41.218966 |
| 2012-10-02 04:00:00 | 0 | 9.439573 | -6.712543 | -2.292124 | 3.206393 | 5.276201 | 5.718478 | 6.576003 | 7.203672 | 8.176026 | ... | 11.509308 | 12.074734 | 12.427330 | 13.552448 | 14.515979 | 15.536160 | 17.744162 | 22.566557 | 27.379155 | 53.829421 |
5 rows × 23 columns
# Calcular cobertura y área de cada intervalo
# ==============================================================================
for interval in intervals:
observed_coverage = calculate_coverage(
y_true = data.loc[end_validation:, 'users'],
lower_bound = predictions[f"p_{interval[0]}"],
upper_bound = predictions[f"p_{interval[1]}"]
)
observed_area = (predictions[f"p_{interval[1]}"] - predictions[f"p_{interval[0]}"]).sum()
observed_coverages.append(100 * observed_coverage)
observed_areas.append(observed_area)
results = pd.DataFrame({
'Interval': intervals,
'Nominal coverage': [interval[1] - interval[0] for interval in intervals],
'Observed coverage': observed_coverages,
'Area': observed_areas
})
results.round(1)
| Interval | Nominal coverage | Observed coverage | Area | |
|---|---|---|---|---|
| 0 | [2.5, 97.5] | 95.0 | 96.7 | 163585.0 |
| 1 | [5, 95] | 90.0 | 93.4 | 129723.9 |
| 2 | [10, 90] | 80.0 | 86.4 | 95072.2 |
| 3 | [15, 85] | 70.0 | 79.8 | 73890.3 |
| 4 | [20, 80] | 60.0 | 74.1 | 58018.5 |
| 5 | [30, 70] | 40.0 | 51.1 | 34447.4 |
| 6 | [35, 65] | 30.0 | 40.6 | 24832.8 |
| 7 | [40, 60] | 20.0 | 26.5 | 16087.9 |
| 8 | [45, 55] | 10.0 | 12.5 | 8026.3 |
Predicción bootstraping, cuantiles y distribución¶
En las secciones anteriores se ha mostrado el uso del proceso de backtesting para estimar el intervalo de predicción a lo largo de un periodo de tiempo determinado. El objetivo es imitar el comportamiento del modelo en producción ejecutando predicciones a intervalos regulares, actualizando incrementalmente los datos de entrada.
También es posible ejecutar una única predicción que estime N steps por delante sin pasar por todo el proceso de backtesting. En estos casos, skforecast ofrece cuatro métodos diferentes: predict_bootstrapping, predict_interval, predict_quantile y predict_dist. Para información detallada sobre estos métodos, consulte la documentación.
Conformal Prediction¶
Conformal prediction engloba un conjunto de técnicas que permiten generar intervalos de predicción garantizando una determinada cobertura. Se basa en combinar las predicciones puntuales de un modelo de forecasting con sus residuales históricos (diferencias entre predicciones previas y valores observados). Estos residuales permiten estimar la incertidumbre en la predicción y ajustar la amplitud del intervalo entorno a la predicción puntual. Skforecast utiliza Split Conformal Prediction (SCP).
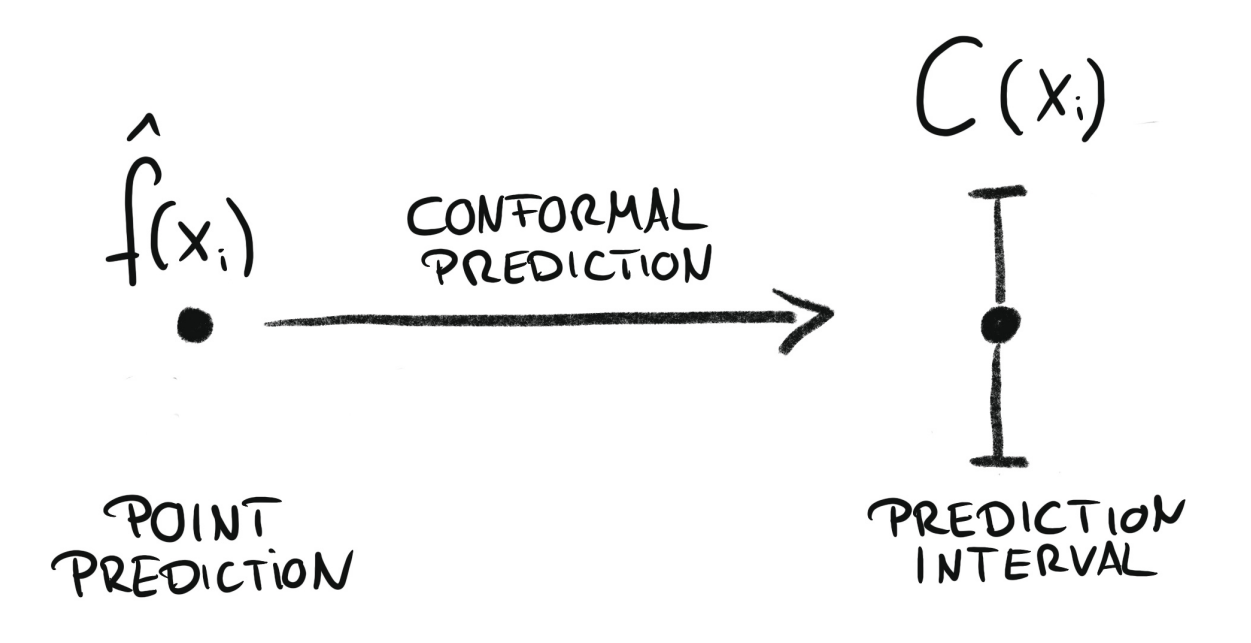
Conformal regression convierte las predicciones puntuales en intervalos de predicción. Fuente: Introduction To Conformal Prediction With Python: A Short Guide For Quantifying Uncertainty Of Machine Learning Models
by Christoph Molnar https://leanpub.com/conformal-prediction
Animación del proceso de predicción conformal probabilística.
Este método se puede utilizar también para calibrar los intervalos de predicción generados por otros métodos, como la regresión cuantílica o bootstrapping. En estos casos, el método conformal ajusta los intervalos para garantizar que sigan siendo válidos con respecto a una determinada de cobertura.
⚠️ Warning
Existen varios métodos de conformal prediction bien establecidos, cada uno con sus propias características y suposiciones. Sin embargo, cuando se aplican al forecasting de series temporales, sus garantías de cobertura solo son válidas para predicciones de un paso adelante (single-step). Para predicciones de varios pasos adelante (multi-step), la cobertura no está garantizada. Skforecast implementa Split Conformal Prediction (SCP) debido a su balance entre complejidad y eficacia.
Se realiza un proceso de backtesting para estimar los intervalos de predicción para el conjunto de test, esta vez utilizando el método conformal. Dado que los residuos outsample ya están almacenados en el objeto forecaster, el argumento use_in_sample_residuals se establece en False, y use_binned_residuals se establece en True para permitir intervalos adaptativos.
# Backtesting con intervalos en los datos de test usando residuos out-sample
# ==============================================================================
cv = TimeSeriesFold(steps = 24, initial_train_size = len(data.loc[:end_validation]))
metric, predictions = backtesting_forecaster(
forecaster = forecaster,
y = data['users'],
exog = data[exog_features],
cv = cv,
metric = 'mean_absolute_error',
interval = 0.8, # 80% prediction interval
interval_method = 'conformal',
use_in_sample_residuals = False, # Use out-sample residuals
use_binned_residuals = True, # Adaptive conformal
)
predictions.head(3)
0%| | 0/19 [00:00<?, ?it/s]
| fold | pred | lower_bound | upper_bound | |
|---|---|---|---|---|
| 2012-10-02 00:00:00 | 0 | 60.399647 | 32.785300 | 88.013993 |
| 2012-10-02 01:00:00 | 0 | 17.617464 | 8.692943 | 26.541985 |
| 2012-10-02 02:00:00 | 0 | 9.002365 | 0.077843 | 17.926886 |
# Gráfico intervalos
# ==============================================================================
plot_predicted_intervals(
predictions = predictions,
y_true = data_test,
target_variable = "users",
xaxis_title = "Date time",
yaxis_title = "users",
)
# Cobertura del intervalo en los datos de test
# ==============================================================================
coverage = calculate_coverage(
y_true = data.loc[end_validation:, 'users'],
lower_bound = predictions["lower_bound"],
upper_bound = predictions["upper_bound"]
)
print(f"Predicted interval coverage: {round(100 * coverage, 2)} %")
# Area del intervalo
# ==============================================================================
area = (predictions["upper_bound"] - predictions["lower_bound"]).sum()
print(f"Area of the interval: {round(area, 2)}")
Predicted interval coverage: 78.29 % Area of the interval: 64139.13
Los intervalos obtenidos muestran una cobertura ligeramente inferior a la 80% esperada, pero cercana a ella.
Regresión cuantílica¶
A diferencia de los modelos de regresión más comunes, que pretende estimar la media de la variable respuesta dados ciertos valores de las variables predictoras, la regresión cuantílica tiene como objetivo estimar los cuantiles condicionales de la variable respuesta. Para una función de distribución continua, el cuantil $\alpha$ $Q_{\alpha}(x)$ se define como el valor tal que la probabilidad de que $Y$ sea menor que $Q_{\alpha}(x)$ es, para un determinado $X=x$, igual a $\alpha$. Por ejemplo, el 36% de los valores de la población son inferiores al cuantil $Q=0,36$. El cuantil más conocido es el cuantil 50%, más comúnmente conocido como mediana.
Al combinar las predicciones de dos modelos de regresión cuantílica, es posible construir un intervalo donde, cada modelo, estima uno de los límites del intervalo. Por ejemplo, los modelos obtenidos para $Q = 0.1$ y $Q = 0.9$ generan un intervalo de predicción del 80% (90% - 10% = 80%).
Son varios los algoritmos de machine learning capaces de modelar cuantiles. Algunos de ellos son:
Así como el error cuadrático se utiliza como función de coste para entrenar modelos que predicen el valor medio, se necesita una función de coste específica para entrenar modelos que predicen cuantiles. La función utilizada con más frecuencia para la regresión de cuantiles se conoce como pinball:
$$\text{pinball}(y, \hat{y}) = \frac{1}{n_{\text{muestras}}} \sum_{i=0}^{n_{\text{muestras}}- 1} \alpha \max(y_i - \hat{y}_i, 0) + (1 - \alpha) \max(\hat{y}_i - y_i, 0)$$donde $\alpha$ es el cuantil objetivo, $y$ el valor real e $\hat{y}$ la predicción del cuantil.
Se puede observar que el coste difiere según el cuantil evaluado. Cuanto mayor sea el cuantil, más se penalizan las subestimaciones y menos las sobreestimaciones. Al igual que con MSE y MAE, el objetivo es minimizar sus valores (a menor coste, mejor).
Dos desventajas de la regresión por cuantiles en comparación con el método de bootstrapping son: que cada cuantil necesita su regresor, y que la regresión por cuantiles no está disponible para todos los tipos de modelos de regresión. Sin embargo, una vez entrenados los modelos, la inferencia es mucho más rápida ya que no se necesita un proceso iterativo.
Este tipo de intervalos de predicción se pueden estimar fácilmente utilizando ForecasterDirect.
⚠️ Warning
Los forecasters de tipo ForecasterDirect son más lentos que ForecasterRecursive porque requieren entrenar un modelo por paso. Aunque pueden lograr un mejor rendimiento, su escalabilidad es una limitación importante cuando se necesitan predecir muchos pasos.
# Crear forecasters: uno para cada límite del intervalo
# ==============================================================================
# Los forecasters obtenidos para alpha=0.1 y alpha=0.9 producen un intervalo de
# confianza del 80% (90% - 10% = 80%).
# Forecaster para cuantil 10%
forecaster_q10 = ForecasterDirect(
estimator = LGBMRegressor(
objective = 'quantile',
metric = 'quantile',
alpha = 0.1,
random_state = 15926,
verbose = -1
),
lags = lags,
steps = 24
)
# Forecaster para cuantil 90%
forecaster_q90 = ForecasterDirect(
estimator = LGBMRegressor(
objective = 'quantile',
metric = 'quantile',
alpha = 0.9,
random_state = 15926,
verbose = -1
),
lags = lags,
steps = 24
)
Una vez definidos los forecasters, se realiza una búsqueda bayesiana para encontrar los mejores hiperparámetros para los regresores. Al validar un modelo de regresión cuantílica, es importante utilizar una métrica coherente con el cuantil que se está evaluando. En este caso, se utiliza función de coste pinball. Skforecast proporciona la función create_mean_pinball_loss para calcular la función de coste pinball para un cuantil dado.
# Grid search para los hyper-parámetros y lags de cada quantile-forecaster
# ==============================================================================
def search_space(trial):
search_space = {
'n_estimators' : trial.suggest_int('n_estimators', 100, 500, step=50),
'max_depth' : trial.suggest_int('max_depth', 3, 10, step=1),
'learning_rate' : trial.suggest_float('learning_rate', 0.01, 0.1)
}
return search_space
cv = TimeSeriesFold(steps = 24, initial_train_size = len(data[:end_train]))
results_grid_q10 = bayesian_search_forecaster(
forecaster = forecaster_q10,
y = data.loc[:end_validation, 'users'],
cv = cv,
metric = create_mean_pinball_loss(alpha=0.1),
search_space = search_space,
n_trials = 10
)
results_grid_q90 = bayesian_search_forecaster(
forecaster = forecaster_q90,
y = data.loc[:end_validation, 'users'],
cv = cv,
metric = create_mean_pinball_loss(alpha=0.9),
search_space = search_space,
n_trials = 10
)
0%| | 0/10 [00:00<?, ?it/s]
`Forecaster` refitted using the best-found lags and parameters, and the whole data set:
Lags: [ 1 2 3 23 24 25 167 168 169]
Parameters: {'n_estimators': 450, 'max_depth': 8, 'learning_rate': 0.06499211596098246}
Backtesting metric: 21.106815983551137
0%| | 0/10 [00:00<?, ?it/s]
`Forecaster` refitted using the best-found lags and parameters, and the whole data set:
Lags: [ 1 2 3 23 24 25 167 168 169]
Parameters: {'n_estimators': 400, 'max_depth': 5, 'learning_rate': 0.042560979006008276}
Backtesting metric: 38.44122580678526
Una vez que se han encontrado los mejores hiperparámetros para cada forecaster, se aplica un proceso de backtesting utilizando los datos de test.
predictions
| fold | pred | lower_bound | upper_bound | |
|---|---|---|---|---|
| 2012-10-02 00:00:00 | 0 | 60.399647 | 32.785300 | 88.013993 |
| 2012-10-02 01:00:00 | 0 | 17.617464 | 8.692943 | 26.541985 |
| 2012-10-02 02:00:00 | 0 | 9.002365 | 0.077843 | 17.926886 |
| 2012-10-02 03:00:00 | 0 | 5.306644 | -3.617878 | 14.231165 |
| 2012-10-02 04:00:00 | 0 | 9.439573 | 0.515052 | 18.364095 |
| ... | ... | ... | ... | ... |
| 2012-10-20 19:00:00 | 18 | 339.453856 | 212.650339 | 466.257373 |
| 2012-10-20 20:00:00 | 18 | 252.646392 | 181.902353 | 323.390431 |
| 2012-10-20 21:00:00 | 18 | 208.253216 | 155.026089 | 261.480344 |
| 2012-10-20 22:00:00 | 18 | 174.512264 | 121.285137 | 227.739391 |
| 2012-10-20 23:00:00 | 18 | 141.429278 | 88.202151 | 194.656405 |
456 rows × 4 columns
# Backtesting con datos de test
# ==============================================================================
cv = TimeSeriesFold(steps = 24, initial_train_size = len(data.loc[:end_validation]))
metric_q10, predictions_q10 = backtesting_forecaster(
forecaster = forecaster_q10,
y = data['users'],
cv = cv,
metric = create_mean_pinball_loss(alpha=0.1)
)
metric_q90, predictions_q90 = backtesting_forecaster(
forecaster = forecaster_q90,
y = data['users'],
cv = cv,
metric = create_mean_pinball_loss(alpha=0.9)
)
predictions = pd.concat([predictions_q10['pred'], predictions_q90['pred']], axis=1)
predictions.columns = ['lower_bound', 'upper_bound']
predictions.head(3)
0%| | 0/19 [00:00<?, ?it/s]
0%| | 0/19 [00:00<?, ?it/s]
| lower_bound | upper_bound | |
|---|---|---|
| 2012-10-02 00:00:00 | 39.108177 | 73.136230 |
| 2012-10-02 01:00:00 | 11.837010 | 32.588538 |
| 2012-10-02 02:00:00 | 4.453201 | 14.346566 |
# Gráfico
# ==============================================================================
fig = go.Figure([
go.Scatter(name='Real value', x=data_test.index, y=data_test['users'], mode='lines'),
go.Scatter(
name='Upper Bound', x=predictions.index, y=predictions['upper_bound'],
mode='lines', marker=dict(color="#444"), line=dict(width=0), showlegend=False
),
go.Scatter(
name='Lower Bound', x=predictions.index, y=predictions['lower_bound'],
marker=dict(color="#444"), line=dict(width=0), mode='lines',
fillcolor='rgba(68, 68, 68, 0.3)', fill='tonexty', showlegend=False
)
])
fig.update_layout(
title="Real value vs predicted in test data",
xaxis_title="Date time",
yaxis_title="users",
width=800,
height=400,
margin=dict(l=20, r=20, t=35, b=20),
hovermode="x",
legend=dict(orientation="h", yanchor="top", y=1.1, xanchor="left", x=0.001)
)
fig.show()
# Cobertura del intervalo en los datos de test
# ==============================================================================
coverage = calculate_coverage(
y_true = data.loc[end_validation:, 'users'],
lower_bound = predictions["lower_bound"],
upper_bound = predictions["upper_bound"]
)
print(f"Cobertura del intervalo: {round(100 * coverage, 2)} %")
# Area del intervalo
# ==============================================================================
area = (predictions_q90["pred"] - predictions_q10["pred"]).sum()
print(f"Area del intervalo: {round(area, 2)}")
Cobertura del intervalo: 62.28 % Area del intervalo: 109062.92
En este caso de uso, la estrategia de previsión cuantílica no logra una cobertura empírica cercana a la esperada (80%).
Calibración externa de intervalos de predicción¶
Con frecuencia, los intervalos de predicción obtenidos con los diferentes métodos no logran la cobertura deseada porque son demasiado optimistas o pesimistas. Para abordar este problema, skforecast proporciona el transformador ConformalIntervalCalibrator, que se puede utilizar para calibrar los intervalos de predicción obtenidos con otros métodos.
Información de sesión¶
import session_info
session_info.show(html=False)
----- feature_engine 1.9.3 lightgbm 4.6.0 matplotlib 3.10.8 numpy 2.2.6 pandas 2.3.3 plotly 6.5.0 session_info v1.0.1 skforecast 0.20.0 sklearn 1.7.2 ----- IPython 9.8.0 jupyter_client 8.8.0 jupyter_core 5.9.1 jupyterlab 4.5.3 notebook 7.5.3 ----- Python 3.13.11 | packaged by Anaconda, Inc. | (main, Dec 10 2025, 21:28:48) [GCC 14.3.0] Linux-6.14.0-37-generic-x86_64-with-glibc2.39 ----- Session information updated at 2026-02-10 09:59
Instrucciones para citar¶
¿Cómo citar este documento?
Si utilizas este documento o alguna parte de él, te agradecemos que lo cites. ¡Muchas gracias!
Forecasting probabilístico con machine learning por Joaquín Amat Rodrigo y Javier Escobar Ortiz, disponible bajo una licencia Attribution-NonCommercial-ShareAlike 4.0 International (CC BY-NC-SA 4.0 DEED) en https://cienciadedatos.net/documentos/py42-forecasting-probabilistico.html
¿Cómo citar skforecast?
Si utilizas skforecast, te agradecemos mucho que lo cites. ¡Muchas gracias!
Zenodo:
Amat Rodrigo, Joaquin, & Escobar Ortiz, Javier. (2024). skforecast (v0.20.0). Zenodo. https://doi.org/10.5281/zenodo.8382788
APA:
Amat Rodrigo, J., & Escobar Ortiz, J. (2024). skforecast (Version 0.20.0) [Computer software]. https://doi.org/10.5281/zenodo.8382788
BibTeX:
@software{skforecast, author = {Amat Rodrigo, Joaquin and Escobar Ortiz, Javier}, title = {skforecast}, version = {0.20.0}, month = {01}, year = {2026}, license = {BSD-3-Clause}, url = {https://skforecast.org/}, doi = {10.5281/zenodo.8382788} }
¿Te ha gustado el artículo? Tu ayuda es importante
Tu contribución me ayudará a seguir generando contenido divulgativo gratuito. ¡Muchísimas gracias! 😊
Este documento creado por Joaquín Amat Rodrigo y Javier Escobar Ortiz tiene licencia Attribution-NonCommercial-ShareAlike 4.0 International.
Se permite:
-
Compartir: copiar y redistribuir el material en cualquier medio o formato.
-
Adaptar: remezclar, transformar y crear a partir del material.
Bajo los siguientes términos:
-
Atribución: Debes otorgar el crédito adecuado, proporcionar un enlace a la licencia e indicar si se realizaron cambios. Puedes hacerlo de cualquier manera razonable, pero no de una forma que sugiera que el licenciante te respalda o respalda tu uso.
-
No-Comercial: No puedes utilizar el material para fines comerciales.
-
Compartir-Igual: Si remezclas, transformas o creas a partir del material, debes distribuir tus contribuciones bajo la misma licencia que el original.


