Más sobre ciencia de datos en: cienciadedatos.net
- Regresión lineal con Python
- Regresión lineal múltiple con Python
- Regresión logística con Python
- Regularización Ridge, Lasso y Elastic Net con Python
- Machine learning con Python y Scikitlearn
- Árboles de decisión con Python: regresión y clasificación
- Random Forest con Python y Scikit-learn
- Gradient Boosting con Python y Scikit-learn
- Gradient Boosting probabilístico con Python
- Máquinas de Vector Soporte (SVM)
- Redes neuronales con Python
- Análisis de componentes principales PCA
- Clustering
- Detección de anomalías con PCA
- Detección de anomalías con autoencoders
- Detección de anomalías con Gaussian Mixture Models
- Detección de anomalías con Isolation Forest
- Análisis de texto (text mining) con Python
- Reglas de asociación
- Análisis Tweets con Python

Introducción¶
La regresión lineal múltiple es un método estadístico que trata de modelar la relación entre una variable continua y dos o más variables independientes mediante el ajuste de una ecuación lineal. La regresión lineal múltiple permite generar un modelo en el que el valor de la variable dependiente o respuesta ($Y$) se determina a partir de un conjunto de variables independientes llamadas predictores ($X_1$, $X_2$, $X_3$…). Es una extensión de la regresión lineal simple, por lo que es fundamental comprender esta última.
Los modelos de regresión múltiple pueden emplearse para predecir el valor de la variable dependiente o para evaluar la influencia que tienen los predictores sobre ella (esto último se debe analizar con cautela para no malinterpretar causa-efecto).
A lo largo de este documento, se describen de forma progresiva los fundamentos teóricos de la regresión lineal múltiple, los principales aspectos prácticos a tener en cuenta y ejemplos de cómo crear este tipo de modelos en Python.
Modelos de regresión lineal en Python
Dos de las implementaciones de modelos de regresión lineal más utilizadas en Python son: scikit-learn y statsmodels. Aunque ambas están muy optimizadas, Scikit-learn está orientada principalmente a la predicción, por lo que no dispone de apenas funcionalidades que muestren las muchas características del modelo que se deben analizar para hacer inferencia. Statsmodels es mucho más completo en este sentido.
✏️ Note
La regresión lineal múltiple es una extensión de la regresión lineal simple, por lo que es recomendable leer primero Regresión lineal con Python.
Definición matemática¶
El modelo de regresión lineal (Legendre, Gauss, Galton y Pearson) considera que, dado un conjunto de observaciones $\{y_i, x_{i1}, ..., x_{np}\}^{n}_{i=1}$, la media $\mu$ de la variable respuesta $y$ se relaciona de forma lineal con la o las variables regresoras $x_1$ ... $x_p$ acorde a la ecuación:
$$\mu_y = \beta_0 + \beta_1 x_{1} + \beta_2 x_{2} + ... + \beta_p x_{p}$$El resultado de esta ecuación se conoce como la línea de regresión poblacional, y recoge la relación entre los predictores y la media de la variable respuesta.
Otra definición que se encuentra con frecuencia en los libros de estadística es:
$$y_i= \beta_0 + \beta_1 x_{i1} + \beta_2 x_{i2} + ... + \beta_p x_{ip} +\epsilon_i$$En este caso, se está haciendo referencia al valor de $y$ para una observación $i$ concreta. El valor de una observación puntual nunca va a ser exactamente igual al promedio, de ahí que se añada el término de error $\epsilon$.
En ambos casos, la interpretación de los elementos del modelo es la misma:
$\beta_{0}$: es la ordenada en el origen, se corresponde con el valor promedio de la variable respuesta $y$ cuando todos los predictores son cero.
$\beta_{j}$: es el efecto promedio que tiene sobre la variable respuesta el incremento en una unidad de la variable predictora $x_{j}$, manteniéndose constantes el resto de variables. Se conocen como coeficientes parciales de regresión.
$\epsilon$: es el residuo o error, la diferencia entre el valor observado y el estimado por el modelo. Recoge el efecto de todas aquellas variables que influyen en $y$ pero que no se incluyen en el modelo como predictores.
En la gran mayoría de casos, los valores $\beta_0$ y $\beta_j$ poblacionales se desconocen, por lo que, a partir de una muestra, se obtienen sus estimaciones $\hat{\beta}_0$ y $\hat{\beta}_j$. Ajustar el modelo consiste en estimar, a partir de los datos disponibles, los valores de los coeficientes de regresión que maximizan la verosimilitud (likelihood), es decir, los que dan lugar al modelo que con mayor probabilidad puede haber generado los datos observados.
El método empleado con más frecuencia es el ajuste por mínimos cuadrados ordinarios (OLS), que identifica como mejor modelo la recta (o plano si es regresión múltiple) que minimiza la suma de las desviaciones verticales entre cada dato de entrenamiento y la recta, elevadas al cuadrado.
Computacionalmente, estos cálculos son más eficientes si se realizan de forma matricial:
$$\mathbf{y}=\mathbf{X} \mathbf{\beta}+\mathbf{\epsilon}$$$$\mathbf{y}=\begin{bmatrix} y_1\\ y_2\\ ....\\ y_n\end{bmatrix} \ , \ \ \ \mathbf{X}=\begin{bmatrix} 1 & x_{11} & ... & x_{1p}\\ 1 & x_{21} & ... & x_{2p}\\ 1 & ... & ... & ... \\ 1 & x_{n1} & ... &x_{np}\\ \end{bmatrix} \ , \ \ \ \mathbf{\beta}=\begin{bmatrix} \beta_0\\ \beta_1\\ ...\\ \beta_p\end{bmatrix} \ , \ \ \ \mathbf{\epsilon}=\begin{bmatrix} \epsilon_1\\ \epsilon_2\\ ...\\ \epsilon_n\end{bmatrix}$$$$\hat{\mathbf{\beta}} = \underset{\mathbf{\beta}}{\arg\min} (\mathbf{y} - \mathbf{X} \mathbf{\beta})^T(\mathbf{y} - \mathbf{X} \mathbf{\beta})$$Una vez estimados los coeficientes, se pueden obtener las estimaciones de cada observación ($\hat{y}_i$):
$$\hat{y}_i= \hat{\beta}_0 + \hat{\beta}_1 x_{i1} + \hat{\beta}_2 x_{i2} + ... + \hat{\beta}_p x_{ip}$$Finalmente, la estimación de la varianza del modelo ($\hat{\sigma}^2$) se obtiene como:
$$\hat{\sigma}^2 = \frac{\sum^n_{i=1} \hat{\epsilon}_i^2}{n-p-1} = \frac{\sum^n_{i=1} (y_i - \hat{y}_i)^2}{n-p-1}$$donde $n$ es el número de observaciones y $p$ el número de predictores. El denominador $n-p-1$ corresponde a los grados de libertad, que se calculan restando del número de observaciones el número de parámetros estimados (los $p$ coeficientes de los predictores más el intercepto $\beta_0$).
Interpretación del modelo¶
Los principales elementos que hay que interpretar en un modelo de regresión lineal son los coeficientes de los predictores:
$\beta_{0}$ es la ordenada en el origen o intercept, se corresponde con el valor esperado de la variable respuesta $y$ cuando todos los predictores son cero.
$\beta_{j}$ los coeficientes de regresión parcial de cada predictor indican el cambio promedio esperado de la variable respuesta $y$ al incrementar en una unidad de la variable predictora $x_{j}$, manteniéndose constantes el resto de variables.
La magnitud de cada coeficiente parcial de regresión depende de las unidades en las que se mida la variable predictora a la que corresponde, por lo que su magnitud no está asociada con la importancia de cada predictor.
Para poder comparar el impacto que tienen en el modelo cada una de las variables, se emplean los coeficientes parciales estandarizados, que se obtienen al estandarizar (sustraer la media y dividir entre la desviación estándar) las variables predictoras previo ajuste del modelo. En este caso, $\beta_{0}$ se corresponde con el valor esperado de la variable respuesta cuando todos los predictores se encuentran en su valor promedio, y $\beta_{j}$ el cambio promedio esperado de la variable respuesta al incrementar en una desviación estándar la variable predictora $x_{j}$, manteniéndose constantes el resto de variables.
Si bien los coeficientes de regresión suelen ser el primer objetivo de la interpretación de un modelo lineal, existen muchos otros aspectos (significancia del modelo en su conjunto, significancia de los predictores, condición de normalidad...). Estos últimos suelen ser tratados con poca detalle cuando el único objetivo del modelo es realizar predicciones, sin embargo, son muy relevantes si se quiere realizar inferencia, es decir, explicar las relaciones entre los predictores y la variable respuesta. A lo largo de este documento se irán introduciendo cada uno de ellos.
Significado de "lineal"
El término "lineal" en los modelos de regresión hace referencia al hecho de que los parámetros se incorporan en la ecuación de forma lineal, no a que necesariamente la relación entre cada predictor y la variable respuesta tenga que seguir un patrón lineal.
La siguiente ecuación muestra un modelo lineal en el que el predictor $x_1$ no es lineal respecto a y:
$$y = \beta_0 + \beta_1x_1 + \beta_2log(x_1) + \epsilon$$En contraposición, el siguiente no es un modelo lineal:
$$y = \beta_0 + \beta_1x_1^{\beta_2} + \epsilon$$En ocasiones, algunas relaciones no lineales pueden transformarse de forma que se pueden expresar de manera lineal:
$$y = \beta_0x_1^{\beta_1}\epsilon $$$$log(y)=log(\beta_0) + \beta_1log(x_1) + log(\epsilon)$$Bondad de ajuste del modelo¶
Una vez ajustado el modelo, es necesario verificar su utilidad ya que, aun siendo la línea que mejor se ajusta a las observaciones de entre todas las posibles, puede tener un gran error. Las métricas más utilizadas para medir la calidad del ajuste son: el error estándar de los residuos y el coeficiente de determinación $R^2$.
Error estándar de los residuos (Residual Standard Error, RSE)
Mide la desviación típica (estándar) de los residuos, es decir, proporciona una estimación de la dispersión promedio de las observaciones alrededor de la recta (o hiperplano en regresión múltiple) de regresión. Tiene las mismas unidades que la variable respuesta. Una forma de saber si el valor del RSE es elevado consiste en dividirlo entre el valor medio de la variable respuesta, obteniendo así un porcentaje de la desviación relativa.
$$RSE = \sqrt{\frac{1}{n-p-1}RSS} = \sqrt{\frac{1}{n-p-1}\sum^n_{i=1}(y_i - \hat{y}_i)^2}$$donde $RSS$ es la suma de cuadrados de los residuos (Residual Sum of Squares). El denominador $n-p-1$ corresponde a los grados de libertad del modelo, siendo $n$ el número de observaciones y $p$ el número de predictores. En modelos lineales simples, dado que hay un único predictor, $p=1$ y $(n-p-1) = (n-2)$.
Coeficiente de determinación $R^2$
$R^2$ describe la proporción de varianza de la variable respuesta explicada por el modelo y relativa a la varianza total. Su valor está acotado entre 0 y 1. Al ser adimensional, presenta la ventaja frente al RSE de ser más fácil de interpretar.
$$R^{2}=\frac{\text{Suma de cuadrados totales - Suma de cuadrados residuales}}{\text{Suma de cuadrados totales}}=$$$$1-\frac{\text{Suma de cuadrados residuales}}{\text{Suma de cuadrados totales}} = $$$$1-\frac{\sum(y_i-\hat{y}_i)^2}{\sum(y_i-\overline{y})^2}$$En los modelos lineales múltiples, cuantos más predictores se incluyan en el modelo, mayor es el valor de $R^2$. Esto es así ya que, por poco que sea, cada predictor va a explicar una parte de la variabilidad observada en $y$. Es por esto que $R^2$ no puede utilizarse para comparar modelos con distinto número de predictores.
$R^2_{ajustado}$ introduce una penalización al valor de $R^2$ por cada predictor que se añade al modelo. El valor de la penalización depende del número de predictores utilizados y del tamaño de la muestra, es decir, del número de grados de libertad. Cuanto mayor es el tamaño de la muestra, más predictores se pueden incorporar en el modelo. $R^2_{ajustado}$ permite encontrar el mejor modelo, aquel que consigue explicar mejor la variabilidad de $y$ con el menor número de predictores.
$$R^{2}_{ajustado} = 1 - \frac{RSS}{SST}\times\frac{n-1}{n-p-1} = R^{2} - (1-R^{2})\frac{p}{n-p-1} = 1-\frac{RSS/(n-p-1)}{SST/(n-1)}$$siendo $RSS$ la suma de cuadrados residuales (Residual Sum of Squares), $SST$ la variabilidad total de $y$ (Sum of Squares Total), $n$ el tamaño de la muestra y $p$ el número de predictores introducidos en el modelo.
Significancia del modelo F-test¶
Uno de los primeros resultados que hay que evaluar al ajustar un modelo es el resultado del test de significancia $F$. Este contraste responde a la pregunta de si el modelo en su conjunto es capaz de predecir la variable respuesta mejor de lo esperado por azar, o lo que es equivalente, si al menos uno de los predictores que forman el modelo contribuye de forma significativa. Para realizar este contraste se compara la suma de residuos cuadrados del modelo de interés con la del modelo sin predictores, formado únicamente por la media (también conocido como suma de cuadrados corregidos por la media, $TSS$).
$$F = \frac{(TSS - RSS)/p}{RSS/(n-p-1)}$$Con frecuencia, la hipótesis nula y alternativa de este test se describen como:
$H_0$: $\beta_1$ = ... = $\beta_{p} = 0$
$H_a$: al menos un $\beta_i \neq 0$
Si el test $F$ resulta significativo, implica que el modelo es útil, pero no que sea el mejor. Podría ocurrir que alguno de sus predictores no fuese necesario.
Significancia de los predictores¶
En la mayoría de casos, aunque el estudio de regresión se aplica a una muestra, el objetivo último es obtener un modelo lineal que explique la relación entre las variables en toda la población. Esto significa que, el modelo generado, es una estimación de la relación poblacional a partir de la relación que se observa en la muestra y, por lo tanto, está sujeta a variaciones. Para cada uno de los coeficientes de la ecuación de regresión lineal ($\beta_j$) se puede calcular su significancia (p-value) y su intervalo de confianza. El test estadístico más empleado es el t-test (existen alternativas no paramétricas).
El test de significancia para los coeficientes ($\beta_j$) del modelo lineal considera como hipótesis:
$H_0$: el predictor $x_j$ no contribuye al modelo ($\beta_j = 0$), en presencia del resto de predictores.
$H_a$: el predictor $x_j$ sí contribuye al modelo ($\beta_j \neq 0$), en presencia del resto de predictores. En el caso de regresión lineal simple, se puede interpretar también como que sí existe relación lineal entre ambas variables por lo que la pendiente del modelo es distinta de cero $\beta_j \neq 0$.
Cálculo del estadístico T y del p-value:
$$t = \frac{\hat{\beta}_j}{se(\hat{\beta}_j)}$$donde
$$SE(\hat{\beta}_j)^2 = \frac{\sigma^2}{(1-R^2_j)\sum^n_{i=1}(x_{ji} -\overline{x}_j)^2}$$siendo $R^2_j$ el coeficiente de determinación de la regresión de $x_j$ sobre el resto de predictores.
La varianza del error $\sigma^2$ se estima a partir del Residual Standard Error (RSE), que puede entenderse como la desviación típica de los residuos. Para regresión lineal múltiple con $p$ predictores, RSE equivale a:
$$RSE = \sqrt{\frac{1}{n-p-1}RSS} = \sqrt{\frac{1}{n-p-1}\sum^n_{i=1}(y_i -\hat{y}_i)^2}$$Grados de libertad (df) = número observaciones - número predictores - 1 = $n-p-1$
p-value = P(|t| > valor calculado de t)
Intervalos de confianza
$$\hat{\beta}_j \pm t_{df}^{\alpha/2} SE(\hat{\beta}_j)$$Cuanto menor es el número de observaciones $n$, menor la capacidad para calcular el error estándar del modelo. Como consecuencia, la exactitud de los coeficientes de regresión estimados se reduce. Esto tiene importancia sobretodo en la regresión múltiple.
En los modelos generados con statsmodels se devuelve, junto con el valor de los coeficientes de regresión, el valor del estadístico $t$ obtenido para cada uno, los p-value y los intervalos de confianza correspondientes. Esto permite saber, además de las estimaciones, si son significativamente distintos de 0, es decir, si contribuyen al modelo.
Para que los cálculos descritos anteriormente sean válidos, se tiene que asumir que los residuos son independientes y que se distribuyen de forma normal con media 0 y varianza $\sigma^2$. Cuando la condición de normalidad no se satisface, existe la posibilidad de recurrir a los test de permutación para calcular significancia (p-value) y al bootstrapping para calcular intervalos de confianza.
Variables categóricas como predictores¶
Cuando se introduce una variable categórica como predictor, un nivel se considera el de referencia (normalmente codificado como 0) y el resto de niveles se comparan con él. En el caso de que el predictor categórico tenga más de dos niveles, se generan lo que se conoce como variables dummy o one-hot-encodding, que son variables creadas para cada uno de los niveles del predictor categórico y que pueden tomar el valor de 0 o 1. Cada vez que se emplee el modelo para predecir un valor, solo una variable dummy por predictor adquiere el valor 1 (la que coincida con el valor que adquiere el predictor en ese caso) mientras que el resto se consideran 0. El valor del coeficiente parcial de regresión $\beta_j$ de cada variable dummy indica el porcentaje promedio en el que influye dicho nivel sobre la variable dependiente $y$ en comparación con el nivel de referencia de dicho predictor.
La idea de variables dummy se entiende mejor con un ejemplo. Supóngase un modelo en el que la variable respuesta peso se predice en función de la altura y nacionalidad del sujeto. La variable nacionalidad es cualitativa con 3 niveles (americana, europea y asiática). A pesar de que el predictor inicial es nacionalidad, se crea una variable nueva por cada nivel, cuyo valor puede ser 0 o 1. De tal forma que la ecuación del modelo completo es:
$$peso = \beta_0 + \beta_1altura + \beta_2americana + \beta_3europea + \beta_4asiatica$$Si el sujeto es europeo, las variables dummy americana y asiática se consideran 0, de forma que el modelo para este caso se queda en:
$$peso = \beta_0 + \beta_1altura + \beta_3europea$$Predicción¶
Una vez generado un modelo válido, es posible predecir el valor de la variable respuesta $y$ para nuevos valores de las variables predictoras $x$. Es importante tener en cuenta que las predicciones deben, a priori, limitarse al rango de valores dentro del que se encuentran las observaciones con las que se ha entrenado el modelo. Esto es importante puesto que, aunque los modelos lineales pueden extrapolarse, solo en esta región se tiene certeza de que se cumplen las condiciones para que el modelo sea válido. Para calcular las predicciones, se emplea la ecuación generada por regresión.
$$\hat{y}_i= \hat{\beta}_0 + \hat{\beta}_1 x_{i1} + \hat{\beta}_2 x_{i2} + ... + \hat{\beta}_p x_{ip}$$Dado que el modelo se ha obtenido a partir de una muestra, las estimaciones de los coeficientes de regresión tienen un error asociado y, por lo tanto, también lo tienen los valores de las predicciones generadas con ellos. Existen dos formas de medir la incertidumbre asociada con una predicción:
- Intervalos de confianza: intervalo del valor promedio esperado de la variable respuesta $y$ para un determinado valor $\mathbf{x}_k$ de los predictores. Refleja la incertidumbre en la estimación de la media de la población.
- Intervalos de predicción: intervalo del valor esperado de una nueva observación individual de la variable respuesta $y$ para un determinado valor $\mathbf{x}_k$ de los predictores. Refleja tanto la incertidumbre en la estimación de la media como la variabilidad inherente de las observaciones individuales.
Si bien ambos parecen similares, la diferencia fundamental es que los intervalos de confianza estiman la incertidumbre del valor promedio de $y$ para un determinado $\mathbf{x}$, mientras que los intervalos de predicción estiman la incertidumbre de una observación individual. Como las observaciones individuales tienen variabilidad adicional (capturada por el término $1$ dentro de la raíz), los intervalos de predicción son siempre más amplios que los de confianza.
En el caso de regresión lineal simple (un solo predictor), estas fórmulas se simplifican a:
Intervalo de confianza (regresión simple): $$\hat{y} \pm t_{\alpha / 2, n - 2} \sqrt{MSE \left(\frac{1}{n} + \frac{(x_k - \bar{x})^2}{\sum(x_i - \bar{x})^2} \right)}$$
Intervalo de predicción (regresión simple): $$\hat{y} \pm t_{\alpha / 2, n - 2} \sqrt{MSE \left(1 + \frac{1}{n} + \frac{(x_k - \bar{x})^2}{\sum (x_i - \bar{x})^2} \right)}$$
Una característica que deriva de la forma en que se calcula el margen de error en los intervalos de confianza y predicción, es que el intervalo se ensancha a medida que los valores de $x$ se aproximan a los extremos del rango observado.
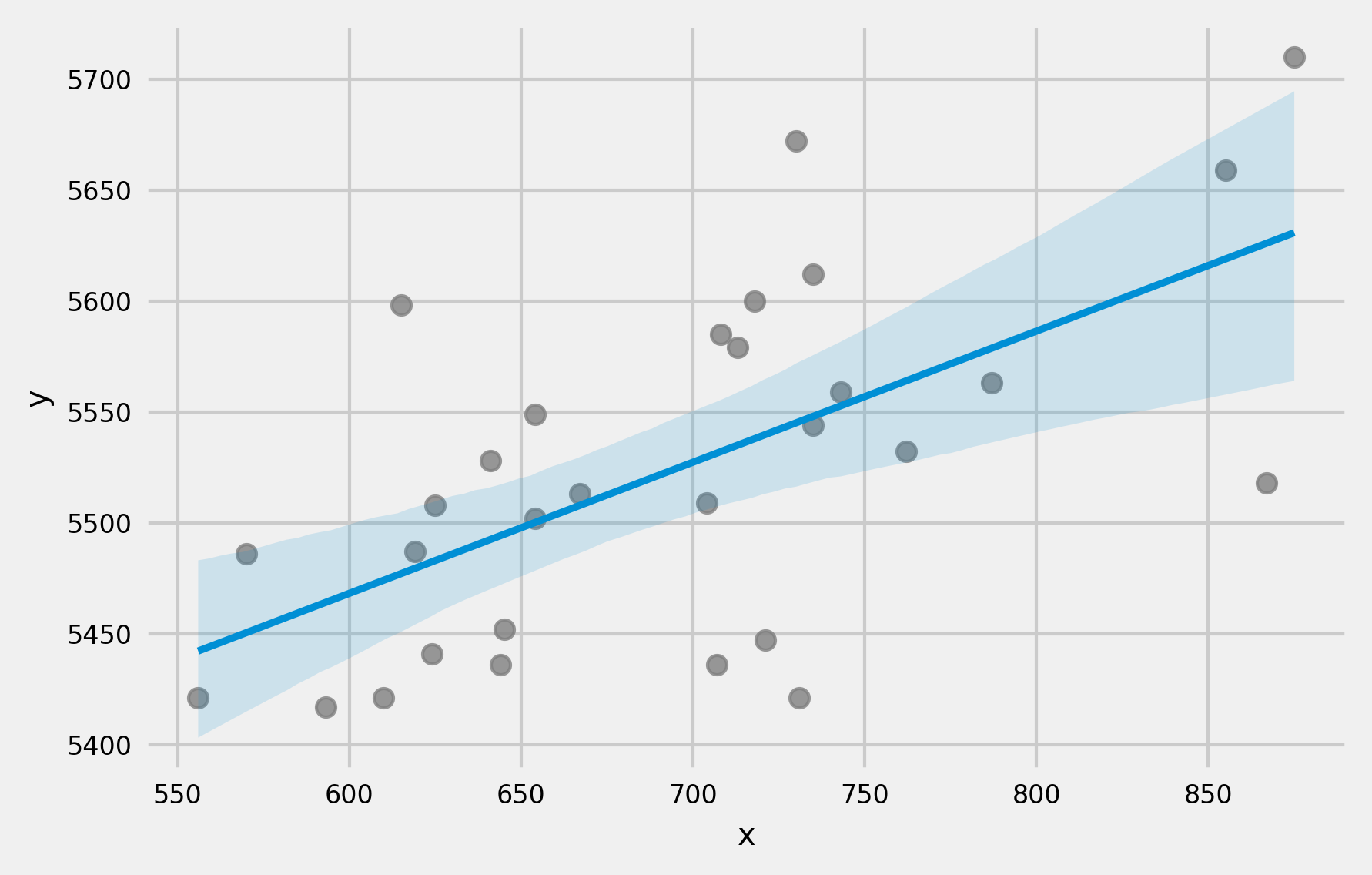
¿Por qué ocurre esto? Prestando atención a la ecuación del error estándar del intervalo de confianza, el numerador contiene el término $(x_k - \bar{x})^2$ (lo mismo ocurre para el intervalo de predicción).
$$\sqrt{MSE \left(\frac{1}{n} + \frac{(x_k - \bar{x})^2}{\sum^n_{i=1}(x_i - \bar{x})^2} \right)}$$Este término se corresponde con la diferencia al cuadrado entre el valor $x_k$ para el que se hace la predicción y la media $\bar{x}$ de los valores observados del predictor $x$. Cuanto más se aleje $x_k$ de $\bar{x}$ mayor es el numerador y por lo tanto el error estándar. Esto se debe a que tenemos más información (observaciones) cerca de la media de nuestros datos, por lo que las predicciones son más precisas en esa región.
Validación del modelo¶
Una vez seleccionado el mejor modelo que se puede crear con los datos disponibles, se tiene que comprobar su capacidad prediciendo nuevas observaciones que no se hayan empleado para entrenarlo, de este modo se verifica si el modelo se puede generalizar. Una estrategia comúnmente empleada es dividir aleatoriamente los datos en dos grupos, ajustar el modelo con el primer grupo y estimar la precisión de las predicciones con el segundo.
El tamaño adecuado de las particiones depende en gran medida de la cantidad de datos disponibles y la seguridad que se necesite en la estimación del error, 80%-20% suele dar buenos resultados.
Condiciones para la regresión lineal múltiple¶
Para que un modelo de regresión lineal por mínimos cuadrados, y las conclusiones derivadas de él, sean completamente válidas, se deben verificar que se cumplen las asunciones sobre las que se basa su desarrollo matemático. En la práctica, rara vez se cumplen, o se puede demostrar que se cumplen todas, sin embargo esto no significa que el modelo no sea útil. Lo importante es ser consciente de ellas y del impacto que esto tiene en las conclusiones que se extraen del modelo.
No colinealidad o multicolinealidad:
En los modelos lineales múltiples, los predictores deben ser independientes, no debe de haber colinealidad entre ellos. La colinealidad ocurre cuando un predictor está linealmente relacionado con uno o varios de los otros predictores del modelo. Como consecuencia de la colinealidad, no se puede identificar de forma precisa el efecto individual que tiene cada predictor sobre la variable respuesta, lo que se traduce en un incremento de la varianza de los coeficientes de regresión estimados hasta el punto de que resulta imposible establecer su significancia estadística. Además, pequeños cambios en los datos, provocan grandes cambios en las estimaciones de los coeficientes. Si bien la colinealidad propiamente dicha existe solo si el coeficiente de correlación simple o múltiple entre predictores es 1, cosa que raramente ocurre en la realidad, es frecuente encontrar la llamada casi-colinealidad o multicolinealidad no perfecta.
No existe un método estadístico concreto para determinar la existencia de colinealidad o multicolinealidad entre los predictores de un modelo de regresión, sin embargo, se han desarrollado numerosas reglas prácticas que tratan de determinar en qué medida afectan al modelo. Los pasos recomendados a seguir son:
Si el coeficiente de determinación $R^2$ es alto pero ninguno de los predictores resulta significativo, hay indicios de colinealidad.
Crear una matriz de correlación en la que se calcula la relación lineal entre cada par de predictores. Es importante tener en cuenta que, a pesar de no obtenerse ningún coeficiente de correlación alto, no está asegurado que no exista multicolinealidad. Se puede dar el caso de tener una relación lineal casi perfecta entre tres o más variables y que las correlaciones simples entre pares de estas mismas variables no sean mayores que 0.5.
Generar un modelo de regresión lineal simple entre cada uno de los predictores frente al resto. Si en alguno de los modelos el *coeficiente de determinación $R^2$ es alto, estaría señalando a una posible colinealidad.
Tolerancia (TOL) y Factor de Inflación de la Varianza (VIF). Se trata de dos parámetros que vienen a cuantificar lo mismo (uno es el inverso del otro). El VIF de cada predictor se calcula según la siguiente fórmula:
Donde $R^2_j$ se obtiene de la regresión del predictor $X_j$ sobre los otros predictores. El $VIF$ cuantifica cuánto aumenta la varianza de un coeficiente de regresión debido a la colinealidad. Esta es la opción más recomendada para detectar multicolinealidad, los límites de referencia que se suelen emplear son:
VIF = 1: ausencia total de colinealidad
1 < VIF < 5: la regresión puede verse afectada por cierta colinealidad.
5 < VIF < 10: la regresión puede verse altamente afectada por cierta colinealidad.
El término tolerancia es $\frac{1}{VIF}$ por lo que los límites recomendables están entre 1 y 0.1.
En caso de encontrar colinealidad entre predictores, hay dos posibles soluciones. La primera es excluir uno de los predictores problemáticos intentando conservar el que, a juicio del investigador, está influyendo realmente en la variable respuesta. Esta medida no suele tener mucho impacto en el modelo en cuanto a su capacidad predictiva ya que, al existir colinealidad, la información que aporta uno de los predictores es redundante en presencia del otro. La segunda opción consiste en combinar las variables colineales en un único predictor, aunque con el riesgo de perder su interpretación.
Cuando se intenta establecer relaciones causa-efecto, la colinealidad puede llevar a conclusiones muy erróneas, haciendo creer que una variable es la causa cuando, en realidad, es otra la que está influenciando sobre ese predictor.
Relación lineal entre los predictores numéricos y la variable respuesta
Cada predictor numérico tiene que estar linealmente relacionado con la variable respuesta $y$ mientras los demás predictores se mantienen constantes, de lo contrario no se deben introducir en el modelo. La forma más recomendable de comprobarlo es representando los residuos del modelo frente a cada uno de los predictores. Si la relación es lineal, los residuos se distribuyen de forma aleatoria en torno a cero. Estos análisis son solo aproximados, ya que no hay forma de saber si realmente la relación es lineal cuando el resto de predictores se mantienen constantes.
Distribución normal de los residuos
Los residuos se deben distribuir de forma normal con media cero. Para comprobarlo se recurre a histogramas, a los cuantiles normales o a test de hipótesis de normalidad.
Varianza constante de la variable respuesta (homocedasticidad)
La varianza de la variable respuesta debe ser constante en todo el rango de los predictores. Para comprobarlo suelen representarse los residuos del modelo frente a cada predictor. Si la varianza es constante, se distribuyen de forma aleatoria manteniendo una misma dispersión y sin ningún patrón específico. Una distribución cónica es un claro identificador de falta de homocedasticidad. También se puede recurrir a contrastes de homocedasticidad como el test de Breusch-Pagan.
La razón de esta condición reside en los siguiente: los modelos lineales asumen que la variable respuesta $y$ sigue una distribución normal, cuya media $\mu$ puede ser modelada en función de otras variables (predictores) y cuya varianza $\sigma$ se calcula mediante una constante de dispersión y una función $\nu(\mu)$. Esto último significa que, la varianza, no se modelan directamente en función de las variables predictoras sino de forma indirecta a través de su relación con la media y es un valor único.
$$\hat{\sigma}^2 = \frac{\sum^n_{i=1} \hat{\epsilon}_i^2}{n-p-1} = \frac{\sum^n_{i=1} (y_i - \hat{y}_i)^2}{n-p-1}$$¿Qué impacto puede tener esto en la práctica? Si bien las estimación de la media obtenidas por estos modelos son buenas, no lo son tanto las incertidumbres asociadas y, en consecuencia, los intervalos de confianza y de predicción que se puedan calcular.
No autocorrelación (Independencia)
Los valores de cada observación son independientes de los otros. Esto es especialmente importante de comprobar cuando se trabaja con mediciones temporales. Se recomienda representar los residuos ordenados acorde al tiempo de registro de las observaciones, si existe un cierto patrón hay indicios de autocorrelación. También se puede emplear el test de hipótesis de Durbin-Watson.
Valores atípicos, con alto leverage o influyentes
Es importante identificar observaciones que sean atípicas o que puedan estar influenciando al modelo. La forma más fácil de detectarlas es a través de los residuos (ver más detalles adelante).
Tamaño de la muestra
No se trata de una condición de por sí pero, si no se dispone de suficientes observaciones, predictores que no son realmente influyentes podrían parecerlo. Un recomendación frecuente es que el número de observaciones sea como mínimo entre 10 y 20 veces el número de predictores del modelo.
Parsimonia
Este término hace referencia a que, el mejor modelo, es aquel capaz de explicar con mayor precisión la variabilidad observada en la variable respuesta empleando el menor número de predictores, por lo tanto, con menos asunciones.
La gran mayoría de condiciones se verifican utilizando los residuos, por lo tanto, se suele generar primero el modelo y posteriormente validar las condiciones. De hecho, el ajuste de un modelo debe verse como un proceso iterativo en el que se ajusta el modelo, se evalúan sus residuos y se mejora. Así hasta llegar a un modelo óptimo.
Selección de los predictores¶
A la hora de seleccionar los predictores que deben formar parte del modelo se pueden seguir varios métodos:
- Método jerárquico: basándose en el criterio del analista, se introducen unos predictores determinados en un orden determinado.
Método de entrada forzada: se introducen todos los predictores simultáneamente.
Método paso a paso (stepwise): emplea criterios matemáticos para decidir qué predictores contribuyen significativamente al modelo y en qué orden se introducen. Dentro de este método se diferencias tres estrategias:
Dirección forward: El modelo inicial no contiene ningún predictor, solo el parámetro $\beta_0$. A partir de este se generan todos los posibles modelos introduciendo una sola variable de entre las disponibles. Aquella variable que mejore en mayor medida el modelo se selecciona. A continuación, se intenta incrementar el modelo probando a introducir una a una las variables restantes. Si introduciendo alguna de ellas mejora, también se selecciona. En el caso de que varias lo hagan, se selecciona la que incremente en mayor medida la capacidad del modelo. Este proceso se repite hasta llegar al punto en el que ninguna de las variables que quedan por incorporar mejore el modelo.
Dirección backward: El modelo se inicia con todas las variables disponibles incluidas como predictores. Se prueba a eliminar una a una cada variable, si se mejora el modelo, queda excluida. Este método permite evaluar cada variable en presencia de las otras.
Doble o mixto: Se trata de una combinación de la selección forward y backward. Se inicia igual que el forward pero tras cada nueva incorporación se realiza un test de extracción de predictores no útiles como en el backward. Presenta la ventaja de que, si a medida que se añaden predictores, alguno de los ya presentes deja de contribuir al modelo, se elimina.
El método paso a paso requiere de algún criterio matemático para determinar si el modelo mejora o empeora con cada incorporación o extracción. Existen varios parámetros empelados, de entre los que destacan el $C_p$, $AIC$, $BIC$ y $R2_{\text{ajustado}}$, cada uno de ellos con ventajas e inconvenientes. El método Akaike(AIC) tiende a ser más restrictivo e introducir menos predictores que el R2-ajustado. Para un mismo set de datos, no todos los métodos tienen porque concluir en un mismo modelo.
Es frecuente encontrar ejemplos en los que la selección de predictores se basa en el p-value asociado a cada uno. Si bien este método es sencillo e intuitivo, presenta múltiples problemas: la inflación del error tipo I debida a las comparaciones múltiples, la eliminación de los predictores menos significativos tiende a incrementar la significancia de los otros predictores. Por esta razón, a excepción de casos muy sencillos con pocos predictores, es preferible no emplear los p-values como criterio de selección.
En el caso de variables categóricas, si al menos uno de sus niveles es significativo, se considera que la variable lo es. Cabe mencionar que, si una variable se excluye del modelo como predictor, significa que no aporta información adicional al modelo, pero sí puede estar relacionada con la variable respuesta.
Valores atípicos (outliers)¶
Independientemente de que el modelo se haya podido aceptar, siempre es conveniente identificar si hay algún posible outlier, observación con alto leverage o influyente, puesto que podría estar condicionando en gran medida el modelo. La eliminación de este tipo de observaciones debe de analizarse con detalle y dependiendo de la finalidad del modelo. Si el fin es predictivo, un modelo sin outliers ni observaciones altamente influyentes suele ser capaz de predecir mejor la mayoría de casos. Sin embargo, es muy importante prestar atención a estos valores ya que, de no tratarse de errores de medida, pueden ser los casos más interesantes. El modo adecuado de proceder cuando se sospecha de algún posible valor atípico o influyente es calcular el modelo de regresión incluyendo y excluyendo dicho valor.
Ejemplo¶
Un estudio quiere generar un modelo que permita predecir la esperanza de vida media de los habitantes de una ciudad en función de diferentes variables. Se dispone de información sobre la esperanza de vida media de los habitantes de 50 ciudades, junto con información sociodemográfica de cada una de ellas. En concreto, se conoce: el número de habitantes, nivel de analfabetismo, ingresos, asesinatos, cantidad de universitarios, heladas, área y densidad poblacional.
Librerías¶
Las librerías utilizadas en este documento son:
# Librerías
# ==============================================================================
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import statsmodels.api as sm
import statsmodels.formula.api as smf
from statsmodels.stats.diagnostic import het_breuschpagan
from statsmodels.stats.outliers_influence import variance_inflation_factor
from statsmodels.tools.eval_measures import rmse
from scipy import stats
from sklearn.model_selection import train_test_split
from sklearn.feature_selection import SequentialFeatureSelector
from sklearn.linear_model import LinearRegression
Datos¶
# Descarga de datos
# ==============================================================================
url = (
'https://raw.githubusercontent.com/JoaquinAmatRodrigo/'
'Estadistica-machine-learning-python/master/data/state_x77.csv'
)
datos = pd.read_csv(url, sep=',')
display(datos.info())
datos.head(3)
<class 'pandas.core.frame.DataFrame'> RangeIndex: 50 entries, 0 to 49 Data columns (total 9 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 habitantes 50 non-null int64 1 ingresos 50 non-null int64 2 analfabetismo 50 non-null float64 3 esp_vida 50 non-null float64 4 asesinatos 50 non-null float64 5 universitarios 50 non-null float64 6 heladas 50 non-null int64 7 area 50 non-null int64 8 densidad_pobl 50 non-null float64 dtypes: float64(5), int64(4) memory usage: 3.6 KB
None
| habitantes | ingresos | analfabetismo | esp_vida | asesinatos | universitarios | heladas | area | densidad_pobl | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 3615 | 3624 | 2.1 | 69.05 | 15.1 | 41.3 | 20 | 50708 | 71.290526 |
| 1 | 365 | 6315 | 1.5 | 69.31 | 11.3 | 66.7 | 152 | 566432 | 0.644384 |
| 2 | 2212 | 4530 | 1.8 | 70.55 | 7.8 | 58.1 | 15 | 113417 | 19.503249 |
# División de los datos en train y test
# ==============================================================================
X = datos.drop(columns='esp_vida')
y = datos['esp_vida']
X_train, X_test, y_train, y_test = train_test_split(
X,
y.values.reshape(-1,1),
train_size = 0.8,
random_state = 1234,
shuffle = True
)
Relación entre variables¶
El primer paso a la hora de crear un modelo lineal múltiple es estudiar la relación que existe entre variables. Esta información es crítica a la hora de identificar cuáles pueden ser los mejores predictores para el modelo, y para detectar colinealidad entre predictores. Además, es recomendable analizar la distribución de cada variable.
✏️ Note
Para información más detallada sobre correlación lineal y distribuciones probabilísticas visitar:
# Correlación lineal entre variables numéricas
# ==============================================================================
corr_matrix = datos.corr(method='pearson')
tril = np.tril(np.ones(corr_matrix.shape)).astype(bool)
corr_matrix[tril] = np.nan
corr_matrix_tidy = corr_matrix.stack().reset_index(name='r')
corr_matrix_tidy = corr_matrix_tidy.rename(columns={'level_0': 'variable_1', 'level_1': 'variable_2'})
corr_matrix_tidy = corr_matrix_tidy.dropna()
corr_matrix_tidy['r_abs'] = corr_matrix_tidy['r'].abs()
corr_matrix_tidy = corr_matrix_tidy.sort_values('r_abs', ascending=False).reset_index(drop=True)
corr_matrix_tidy
| variable_1 | variable_2 | r | r_abs | |
|---|---|---|---|---|
| 0 | esp_vida | asesinatos | -0.780846 | 0.780846 |
| 1 | analfabetismo | asesinatos | 0.702975 | 0.702975 |
| 2 | analfabetismo | heladas | -0.671947 | 0.671947 |
| 3 | analfabetismo | universitarios | -0.657189 | 0.657189 |
| 4 | ingresos | universitarios | 0.619932 | 0.619932 |
| 5 | analfabetismo | esp_vida | -0.588478 | 0.588478 |
| 6 | esp_vida | universitarios | 0.582216 | 0.582216 |
| 7 | asesinatos | heladas | -0.538883 | 0.538883 |
| 8 | asesinatos | universitarios | -0.487971 | 0.487971 |
| 9 | ingresos | analfabetismo | -0.437075 | 0.437075 |
| 10 | universitarios | heladas | 0.366780 | 0.366780 |
| 11 | ingresos | area | 0.363315 | 0.363315 |
| 12 | habitantes | asesinatos | 0.343643 | 0.343643 |
| 13 | area | densidad_pobl | -0.341389 | 0.341389 |
| 14 | ingresos | esp_vida | 0.340255 | 0.340255 |
| 15 | universitarios | area | 0.333542 | 0.333542 |
| 16 | habitantes | heladas | -0.332152 | 0.332152 |
| 17 | ingresos | densidad_pobl | 0.329968 | 0.329968 |
| 18 | esp_vida | heladas | 0.262068 | 0.262068 |
| 19 | habitantes | densidad_pobl | 0.246228 | 0.246228 |
| 20 | ingresos | asesinatos | -0.230078 | 0.230078 |
| 21 | asesinatos | area | 0.228390 | 0.228390 |
| 22 | ingresos | heladas | 0.226282 | 0.226282 |
| 23 | habitantes | ingresos | 0.208228 | 0.208228 |
| 24 | asesinatos | densidad_pobl | -0.185035 | 0.185035 |
| 25 | habitantes | analfabetismo | 0.107622 | 0.107622 |
| 26 | esp_vida | area | -0.107332 | 0.107332 |
| 27 | habitantes | universitarios | -0.098490 | 0.098490 |
| 28 | esp_vida | densidad_pobl | 0.091062 | 0.091062 |
| 29 | universitarios | densidad_pobl | -0.088367 | 0.088367 |
| 30 | analfabetismo | area | 0.077261 | 0.077261 |
| 31 | habitantes | esp_vida | -0.068052 | 0.068052 |
| 32 | heladas | area | 0.059229 | 0.059229 |
| 33 | habitantes | area | 0.022544 | 0.022544 |
| 34 | analfabetismo | densidad_pobl | 0.009274 | 0.009274 |
| 35 | heladas | densidad_pobl | 0.002277 | 0.002277 |
# Heatmap matriz de correlaciones
# ==============================================================================
fig, ax = plt.subplots(nrows=1, ncols=1, figsize=(4, 4))
sns.heatmap(
corr_matrix,
annot = True,
cbar = False,
annot_kws = {"size": 8},
vmin = -1,
vmax = 1,
center = 0,
cmap = "viridis",
square = True,
ax = ax
)
ax.set_xticklabels(
ax.get_xticklabels(),
rotation = 45,
horizontalalignment = 'right',
)
ax.tick_params(labelsize = 10)
# Gráfico de distribución para cada variable numérica
# ==============================================================================
# Ajustar número de subplots en función del número de columnas
fig, axes = plt.subplots(nrows=3, ncols=3, figsize=(9, 6))
axes = axes.flat
columnas_numeric = datos.select_dtypes(include=np.number).columns
for i, colum in enumerate(columnas_numeric):
sns.histplot(
data = datos,
x = colum,
stat = "count",
kde = True,
color = (list(plt.rcParams['axes.prop_cycle'])*2)[i]["color"],
ax = axes[i]
)
axes[i].set_title(colum, fontsize = 10)
axes[i].tick_params(labelsize = 8)
axes[i].set_xlabel("")
fig.tight_layout()
plt.subplots_adjust(top = 0.9)
fig.suptitle('Distribución variables numéricas', fontsize = 12);
Del análisis preliminar se pueden extraer las siguientes conclusiones:
Las variables que tienen una mayor relación lineal con la esperanza de vida son: asesinatos (r= -0.78), analfabetismo (r= -0.59) y universitarios (r= 0.58).
Asesinatos y analfabetismo están medianamente correlacionados (r = 0.7) por lo que posiblemente sea redundante introducir ambos predictores en el modelo.
Las variables habitantes, área y densidad poblacional muestran una distribución exponencial, una transformación logarítmica posiblemente haría más normal su distribución.
Ajuste del modelo¶
Se ajusta un primer modelo utilizando todas las variables disponibles como predictores.
✏️ Note
A diferencia de la API de scikit-learn, en la librería de statsmodels, el método fit devuelve un nuevo objeto como resultado en lugar de modificar el objeto existente.
# Ajuste del modelo utilizando el modo fórmula (similar a R)
# ==============================================================================
# Para ajustar el modelo utilizando el modo fórmula, es necesario que los datos
# estén almacenados en un único dataframe.
# datos_train = X_train.assign(esp_vida = y_train).copy()
# modelo = smf.ols(
# formula = 'esp_vida ~ habitantes + ingresos + analfabetismo + asesinatos + universitarios + heladas + area + densidad_pobl',
# data = datos_train
# )
# modelo_res = modelo.fit()
#print(modelo_res.summary())
# Ajuste del modelo utilizando matrices X, y (similar a scikit-learn)
# ==============================================================================
# A la matriz de predictores se le tiene que añadir una columna de 1s
# para el intercept del modelo
X_train = sm.add_constant(X_train, prepend=True).rename(columns={'const':'intercept'})
modelo = sm.OLS(endog=y_train, exog=X_train)
modelo_res = modelo.fit()
print(modelo_res.summary())
OLS Regression Results
==============================================================================
Dep. Variable: y R-squared: 0.760
Model: OLS Adj. R-squared: 0.698
Method: Least Squares F-statistic: 12.28
Date: Sun, 11 Jan 2026 Prob (F-statistic): 1.02e-07
Time: 16:02:09 Log-Likelihood: -42.442
No. Observations: 40 AIC: 102.9
Df Residuals: 31 BIC: 118.1
Df Model: 8
Covariance Type: nonrobust
==================================================================================
coef std err t P>|t| [0.025 0.975]
----------------------------------------------------------------------------------
intercept 70.3069 2.171 32.387 0.000 65.879 74.734
habitantes 9e-05 4.15e-05 2.171 0.038 5.44e-06 0.000
ingresos 0.0002 0.000 0.663 0.512 -0.001 0.001
analfabetismo 0.2330 0.487 0.479 0.635 -0.759 1.225
asesinatos -0.3379 0.057 -5.925 0.000 -0.454 -0.222
universitarios 0.0414 0.027 1.536 0.135 -0.014 0.096
heladas -0.0053 0.004 -1.241 0.224 -0.014 0.003
area -1.101e-06 2.2e-06 -0.501 0.620 -5.58e-06 3.38e-06
densidad_pobl -0.0012 0.001 -1.305 0.202 -0.003 0.001
==============================================================================
Omnibus: 3.407 Durbin-Watson: 2.538
Prob(Omnibus): 0.182 Jarque-Bera (JB): 1.741
Skew: -0.188 Prob(JB): 0.419
Kurtosis: 2.049 Cond. No. 2.07e+06
==============================================================================
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
[2] The condition number is large, 2.07e+06. This might indicate that there are
strong multicollinearity or other numerical problems.
El modelo con todas las variables introducidas como predictores tiene un $R^2$ alto (0.76), es capaz de explicar el 76% de la variabilidad observada en la esperanza de vida. El p-value del modelo es significativo (1.02e-07) por lo que se puede considerar que el modelo predice la variable respuesta mejor de lo que cabría esperar por azar, al menos uno de los coeficientes parciales de regresión es distinto de 0. Muchos de los coeficientes parciales de regresión no son significativos, lo que es un indicativo de que podrían no contribuir al modelo, en presencia del resto.
Selección de los mejores predictores¶
En el momento de escribir este documento, statsmodels no incluye ningún método para la selección recursiva de predictores. Afortunadamente, es relativamente sencillo conseguir una implementación básica.
# Funciones de selección forward y backward para modelos lineales de statsmodels
# ==============================================================================
def forward_selection(
X: pd.DataFrame,
y: pd.Series,
criterio: str='aic',
add_constant: bool=True,
verbose: bool=True
)-> list:
"""
Realiza un procedimiento de selección de variables hacia adelante (forward)
utilizando como criterio de bondad la métrica especificada. El procedimiento
se detiene cuando no es posible mejorar más el modelo añadiendo variables.
Parameters
----------
X: pd.DataFrame
Matriz de predictores
y: pd.Series
Variable respuesta
metrica: str, default='aic'
Métrica utilizada para seleccionar las variables. Debe ser una de las
siguientes opciones: 'aic', 'bic', 'rsquared_adj'.
add_constant: bool, default=True
Si `True` añade una columna de 1s a la matriz de predictores con el
con el nombre de intercept.
verbose: bool, default=True
Si `True` muestra por pantalla los resultados de cada iteración.
Returns
-------
seleccion: list
Lista con las variables seleccionadas.
"""
if add_constant:
X = sm.add_constant(X, prepend=True).rename(columns={'const':'intercept'})
restantes = X.columns.to_list()
seleccion = []
if criterio == 'rsquared_adj':
mejor_metrica = -np.inf
ultima_metrica = -np.inf
else:
mejor_metrica = np.inf
ultima_metrica = np.inf
while restantes:
metricas = []
for candidata in restantes:
seleccion_temp = seleccion + [candidata]
modelo = sm.OLS(endog=y, exog=X[seleccion_temp])
modelo_res = modelo.fit()
metrica = getattr(modelo_res, criterio)
metricas.append(metrica)
if criterio == 'rsquared_adj':
mejor_metrica = max(metricas)
if mejor_metrica > ultima_metrica:
mejor_variable = restantes[np.argmax(metricas)]
else:
break
else:
mejor_metrica = min(metricas)
if mejor_metrica < ultima_metrica:
mejor_variable = restantes[np.argmin(metricas)]
else:
break
seleccion.append(mejor_variable)
restantes.remove(mejor_variable)
ultima_metrica = mejor_metrica
if verbose:
print(f'variables: {seleccion} | {criterio}: {mejor_metrica:.3f}')
return sorted(seleccion)
def backward_selection(
X: pd.DataFrame,
y: pd.Series,
criterio: str='aic',
add_constant: bool=True,
verbose: bool=True
)-> list:
"""
Realiza un procedimiento de selección de variables hacia atrás (backward)
utilizando como criterio de bondad la métrica especificada. El procedimiento
se detiene cuando no es posible mejorar más el modelo eliminando variables.
Parameters
----------
X: pd.DataFrame
Matriz de predictores
y: pd.Series
Variable respuesta
metrica: str, default='aic'
Métrica utilizada para seleccionar las variables. Debe ser una de las
siguientes opciones: 'aic', 'bic', 'rsquared_adj'.
add_constant: bool, default=True
Si `True` añade una columna de 1s a la matriz de predictores con el
con el nombre de intercept.
verbose: bool, default=True
Si `True` muestra por pantalla los resultados de cada iteración.
Returns
-------
seleccion: list
Lista con las variables seleccionadas.
"""
if add_constant:
X = sm.add_constant(X, prepend=True).rename(columns={'const':'intercept'})
# Se inicia con todas las variables como predictores
seleccion = X.columns.to_list()
modelo = sm.OLS(endog=y, exog=X[seleccion])
modelo_res = modelo.fit()
ultima_metrica = getattr(modelo_res, criterio)
mejor_metrica = ultima_metrica
if verbose:
print(f'variables: {seleccion} | {criterio}: {mejor_metrica:.3f}')
while seleccion:
metricas = []
for candidata in seleccion:
seleccion_temp = seleccion.copy()
seleccion_temp.remove(candidata)
modelo = sm.OLS(endog=y, exog=X[seleccion_temp])
modelo_res = modelo.fit()
metrica = getattr(modelo_res, criterio)
metricas.append(metrica)
if criterio == 'rsquared_adj':
mejor_metrica = max(metricas)
if mejor_metrica > ultima_metrica:
peor_variable = seleccion[np.argmax(metricas)]
else:
break
else:
mejor_metrica = min(metricas)
if mejor_metrica < ultima_metrica:
peor_variable = seleccion[np.argmin(metricas)]
else:
break
seleccion.remove(peor_variable)
ultima_metrica = mejor_metrica
if verbose:
print(f'variables: {seleccion} | {criterio}: {mejor_metrica:.3f}')
return sorted(seleccion)
# Selección de variables forward
# ==============================================================================
predictores = forward_selection(
X = X_train,
y = y_train,
criterio = 'aic',
add_constant = False, # Ya se le añadió anteriormente
verbose = True
)
predictores
variables: ['intercept'] | aic: 143.990 variables: ['intercept', 'asesinatos'] | aic: 104.951 variables: ['intercept', 'asesinatos', 'universitarios'] | aic: 102.243 variables: ['intercept', 'asesinatos', 'universitarios', 'habitantes'] | aic: 99.607 variables: ['intercept', 'asesinatos', 'universitarios', 'habitantes', 'heladas'] | aic: 97.043
['asesinatos', 'habitantes', 'heladas', 'intercept', 'universitarios']
# Selección de variables backward
# ==============================================================================
predictores= backward_selection(
X = X_train,
y = y_train,
criterio = 'aic',
add_constant = False, # Ya se le añadió anteriormente
verbose = True
)
predictores
variables: ['intercept', 'habitantes', 'ingresos', 'analfabetismo', 'asesinatos', 'universitarios', 'heladas', 'area', 'densidad_pobl'] | aic: 102.884 variables: ['intercept', 'habitantes', 'ingresos', 'asesinatos', 'universitarios', 'heladas', 'area', 'densidad_pobl'] | aic: 101.179 variables: ['intercept', 'habitantes', 'ingresos', 'asesinatos', 'universitarios', 'heladas', 'densidad_pobl'] | aic: 99.295 variables: ['intercept', 'habitantes', 'asesinatos', 'universitarios', 'heladas', 'densidad_pobl'] | aic: 97.549 variables: ['intercept', 'habitantes', 'asesinatos', 'universitarios', 'heladas'] | aic: 97.043
['asesinatos', 'habitantes', 'heladas', 'intercept', 'universitarios']
Ambas estrategias identifican como mejor modelo el formado por los predictores: 'asesinatos', 'habitantes', 'heladas', 'universitarios' junto con la intersección (intercept).
# Entrenamiento del modelo con las variables seleccionadas
# ==============================================================================
modelo_final = sm.OLS(endog=y_train, exog=X_train[predictores])
modelo_final_res = modelo_final.fit()
print(modelo_final_res.summary())
OLS Regression Results
==============================================================================
Dep. Variable: y R-squared: 0.747
Model: OLS Adj. R-squared: 0.718
Method: Least Squares F-statistic: 25.81
Date: Sun, 11 Jan 2026 Prob (F-statistic): 5.11e-10
Time: 16:02:10 Log-Likelihood: -43.521
No. Observations: 40 AIC: 97.04
Df Residuals: 35 BIC: 105.5
Df Model: 4
Covariance Type: nonrobust
==================================================================================
coef std err t P>|t| [0.025 0.975]
----------------------------------------------------------------------------------
asesinatos -0.3101 0.042 -7.423 0.000 -0.395 -0.225
habitantes 7.437e-05 3.5e-05 2.125 0.041 3.31e-06 0.000
heladas -0.0061 0.003 -2.057 0.047 -0.012 -7.93e-05
intercept 71.0177 1.079 65.845 0.000 68.828 73.207
universitarios 0.0475 0.017 2.772 0.009 0.013 0.082
==============================================================================
Omnibus: 3.043 Durbin-Watson: 2.502
Prob(Omnibus): 0.218 Jarque-Bera (JB): 1.525
Skew: -0.087 Prob(JB): 0.466
Kurtosis: 2.059 Cond. No. 4.66e+04
==============================================================================
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
[2] The condition number is large, 4.66e+04. This might indicate that there are
strong multicollinearity or other numerical problems.
Cada una de las pendientes de un modelo de regresión lineal múltiple (coeficientes parciales de regresión de los predictores) se define del siguiente modo: Si el resto de variables se mantienen constantes, por cada unidad que aumenta el predictor en cuestión, la variable respuesta $Y$ varía en promedio tantas unidades como indica la pendiente. Para este ejemplo, por cada unidad que aumenta el predictor universitarios, la esperanza de vida aumenta en promedio 0.0475 unidades, manteniéndose constantes el resto de predictores.
✏️ Note
¿En qué se diferencia este método de las técnicas existentes en sklearn.feature_selection? Scikit-learn proporciona dos estrategias principales para la selección de predictores:
- Utilizar coeficientes de regressión de cada predictor, en el caso de modelos lineales, u otra métrica de importancia como en los de algoritmos basados en árboles, para eliminar predictores.
- Utilizar una métrica de validación definida por el usuario (mediante partición de validación fija o validación cruzada) para seleccionar los predictores que se eliminan o añaden.
# Selección de predictores con sklearn SequentialFeatureSelector
# ==============================================================================
modelo = LinearRegression()
sfs = SequentialFeatureSelector(
modelo,
n_features_to_select = 'auto',
direction = 'forward',
scoring = 'r2',
cv = 5
)
sfs.fit(X_train, y_train)
sfs.get_feature_names_out().tolist()
['intercept', 'habitantes', 'asesinatos', 'densidad_pobl']
Siguiendo una estrategia de selección secuencial basada en el $R^2$ y con validación cruzada de 5 particiones, los predictores seleccionados son: 'intercept', 'habitantes', 'asesinatos', 'densidad_pobl'.
✏️ Note
Normalmente, los distintos métodos no obtienen resultados equivalentes. Un enfoque eficaz consiste en identificar el subconjunto de predictores en el que coinciden los distintos métodos. Aunque la selección secuencial/recursiva de variables se ha empleado mucho históricamente, nuevos métodos ofrecen características ventajosas. Por ejemplo, es uso de la penalización L1, tambien conocida como Lasso.
Diagnóstico de los resíduos¶
# Residuos del modelo
# ==============================================================================
residuos = modelo_final_res.resid
# prediciones de entrenamiento
# ==============================================================================
prediccion_train = modelo_final_res.predict(X_train[predictores])
Inspección visual
# Gráficos
# ==============================================================================
fig, axes = plt.subplots(nrows=3, ncols=2, figsize=(7, 7))
axes[0, 0].scatter(y_train, prediccion_train, edgecolors=(0, 0, 0), alpha = 0.4)
axes[0, 0].plot([y_train.min(), y_train.max()], [y_train.min(), y_train.max()], 'k--', lw=2)
axes[0, 0].set_title('Valor predicho vs valor real', fontsize=10)
axes[0, 0].set_xlabel('Real')
axes[0, 0].set_ylabel('Predicción')
axes[0, 0].tick_params(labelsize = 7)
axes[0, 1].scatter(list(range(len(y_train))), residuos, edgecolors=(0, 0, 0), alpha=0.4)
axes[0, 1].axhline(y=0, linestyle='--', color='black', lw=2)
axes[0, 1].set_title('Residuos del modelo', fontsize = 10)
axes[0, 1].set_xlabel('id')
axes[0, 1].set_ylabel('Residuo')
axes[0, 1].tick_params(labelsize = 7)
sns.histplot(
data = residuos,
stat = "density",
kde = True,
line_kws = {'linewidth': 1},
alpha = 0.3,
ax = axes[1, 0]
)
axes[1, 0].set_title('Distribución residuos del modelo', fontsize=10)
axes[1, 0].set_xlabel("Residuo")
axes[1, 0].tick_params(labelsize = 7)
sm.qqplot(
residuos,
fit = True,
line = 'q',
ax = axes[1, 1],
color = 'firebrick',
alpha = 0.4,
lw = 2
)
axes[1, 1].set_title('Q-Q residuos del modelo', fontsize=10)
axes[1, 1].tick_params(labelsize=7)
axes[2, 0].scatter(prediccion_train, residuos, edgecolors=(0, 0, 0), alpha=0.4)
axes[2, 0].axhline(y=0, linestyle='--', color='black', lw=2)
axes[2, 0].set_title('Residuos del modelo vs predicción', fontsize=10)
axes[2, 0].set_xlabel('Predicción')
axes[2, 0].set_ylabel('Residuo')
axes[2, 0].tick_params(labelsize=7)
# Se eliminan los axes vacíos
fig.delaxes(axes[2,1])
fig.tight_layout()
plt.subplots_adjust(top=0.9)
fig.suptitle('Diagnóstico residuos', fontsize=12);
Los residuos parecen distribuirse de forma aleatoria en torno a cero, manteniendo aproximadamente la misma variabilidad a lo largo del eje X. Este patrón apunta a que se cumple la normalidad y la homocedasticidad de los residuos.
Test de normalidad
Se comprueba si los residuos siguen una distribución normal empleando dos test estadísticos: Shapiro-Wilk test y D'Agostino's K-squared test. Este último es el que incluye el summary de statsmodels bajo el nombre de Omnibus.
En ambos test, la hipótesis nula considera que los datos siguen una distribución normal, por lo tanto, si el p-value no es inferior al nivel de referencia alpha seleccionado, no hay evidencias para descartar que los datos se distribuyen de forma normal.
# Normalidad de los residuos Shapiro-Wilk test
# ==============================================================================
shapiro_test = stats.shapiro(residuos)
print(f"Test Shapiro-Wilk: estadístico = {shapiro_test[0]}, p-value = {shapiro_test[1]}")
# Normalidad de los residuos D'Agostino's K-squared test
# ==============================================================================
k2, p_value = stats.normaltest(residuos)
print(f"Test D'Agostino's K-squared: estadístico = {k2}, p-value = {p_value}")
Test Shapiro-Wilk: estadístico = 0.9700626181523515, p-value = 0.3615595274049066 Test D'Agostino's K-squared: estadístico = 3.0425718093907816, p-value = 0.21843082505797903
Tanto el análisis gráfico como los test de hipótesis confirman la normalidad.
Homodecasticidad
Al representar los residuos frente a los valores ajustados por el modelo (gráfico "Residuos del modelo vs predicción"), los primeros se distribuyen de forma aleatoria en torno a cero, manteniendo aproximadamente la misma variabilidad a lo largo del eje X.
# Prueba de Breusch-Pagan
# ==============================================================================
lm, lm_p_value, fvalue, f_p_value = het_breuschpagan(residuos, X_train[predictores])
print(f"Estadítico= {fvalue}, p-value = {f_p_value}")
Estadítico= 2.3825683928746826, p-value = 0.07014338688009708
Ni el análisis gráfico ni el test de hipótesis muestran indicios de falta de homocedasticidad.
Multicolinealidad (Inflación de varianza VIF)
# Correlación entre predictores numéricos
# ==============================================================================
corr_matrix = X_train[predictores].corr(method='pearson')
tril = np.tril(np.ones(corr_matrix.shape)).astype(bool)
corr_matrix[tril] = np.nan
corr_matrix_tidy = corr_matrix.stack().reset_index(name='r')
corr_matrix_tidy = corr_matrix_tidy.rename(columns={'level_0': 'variable_1', 'level_1': 'variable_2'})
corr_matrix_tidy = corr_matrix_tidy.dropna()
corr_matrix_tidy['r_abs'] = corr_matrix_tidy['r'].abs()
corr_matrix_tidy = corr_matrix_tidy.sort_values('r_abs', ascending=False).reset_index(drop=True)
corr_matrix_tidy
| variable_1 | variable_2 | r | r_abs | |
|---|---|---|---|---|
| 0 | asesinatos | heladas | -0.549982 | 0.549982 |
| 1 | asesinatos | universitarios | -0.517231 | 0.517231 |
| 2 | heladas | universitarios | 0.448608 | 0.448608 |
| 3 | asesinatos | habitantes | 0.366638 | 0.366638 |
| 4 | habitantes | heladas | -0.228660 | 0.228660 |
| 5 | habitantes | universitarios | -0.195857 | 0.195857 |
# Cálculo del VIF
# ==============================================================================
vif = pd.DataFrame()
vif["variables"] = X_train[predictores].columns
vif["VIF"] = [variance_inflation_factor(X_train[predictores].values, i) for i in range(X_train[predictores].shape[1])]
vif
| variables | VIF | |
|---|---|---|
| 0 | asesinatos | 1.788412 |
| 1 | habitantes | 1.156697 |
| 2 | heladas | 1.515200 |
| 3 | intercept | 78.917401 |
| 4 | universitarios | 1.441241 |
Accorde a la documentación de statsmodels, si el VIF es mayor que 5, entonces el predictor es altamente colineal con los otros predictores.
No hay predictores que muestren una correlación lineal muy alta ni inflación de varianza.
Predicciones¶
Una vez entrenado el modelo, se pueden obtener predicciones para nuevos datos.
# Prediciones del conjunto de test
# ==============================================================================
X_test = sm.add_constant(X_test, prepend=True).rename(columns={'const':'intercept'})
modelo_final_res.predict(X_test[predictores])
37 71.627080 45 70.197194 6 72.102241 44 71.038126 13 70.982884 36 72.470001 8 70.749750 29 71.745882 4 72.255344 49 70.842206 dtype: float64
# Predicciones con intervalo de confianza
# ==============================================================================
# La columna mean contiene la media de la predicción
predicciones = modelo_final_res.get_prediction(exog = X_test[predictores]).summary_frame(alpha=0.05)
predicciones
| mean | mean_se | mean_ci_lower | mean_ci_upper | obs_ci_lower | obs_ci_upper | |
|---|---|---|---|---|---|---|
| 37 | 71.627080 | 0.340585 | 70.935656 | 72.318503 | 69.921765 | 73.332394 |
| 45 | 70.197194 | 0.144249 | 69.904353 | 70.490036 | 68.611071 | 71.783317 |
| 6 | 72.102241 | 0.188786 | 71.718985 | 72.485497 | 70.496964 | 73.707519 |
| 44 | 71.038126 | 0.211805 | 70.608140 | 71.468113 | 69.421055 | 72.655197 |
| 13 | 70.982884 | 0.143523 | 70.691516 | 71.274252 | 69.397032 | 72.568735 |
| 36 | 72.470001 | 0.317495 | 71.825451 | 73.114551 | 70.783148 | 74.156854 |
| 8 | 70.749750 | 0.305186 | 70.130190 | 71.369310 | 69.072286 | 72.427213 |
| 29 | 71.745882 | 0.214940 | 71.309531 | 72.182233 | 70.127107 | 73.364657 |
| 4 | 72.255344 | 0.677814 | 70.879309 | 73.631379 | 70.176041 | 74.334647 |
| 49 | 70.842206 | 0.271753 | 70.290518 | 71.393894 | 69.188607 | 72.495805 |
Error de test¶
# Error de test del modelo
# ==============================================================================
error = rmse(y_test.flatten(), predicciones['mean'])
print(f"El error (rmse) de test es: {error}")
El error (rmse) de test es: 0.5791307323521434
Interpretación¶
El modelo de regresión lineal múltiple
$$Esperanza \ de \ vida =$$$$71.0177 + 7.437^{-05} habitantes - 0.3101 asesinatos + 0.0475universitarios -0.0061 heladas$$es capaz de explicar el 74.7% de la variabilidad observada en la esperanza de vida (R2: 0.747, R2-Adjusted: 0.718). El test F muestra que es significativo (p-value: 5.11e-10). Se satisfacen todas las condiciones para este tipo de regresión múltiple.
Información de sesión¶
import session_info
session_info.show(html=False)
----- matplotlib 3.10.8 numpy 2.2.6 pandas 2.3.3 scipy 1.15.3 seaborn 0.13.2 session_info v1.0.1 sklearn 1.7.2 statsmodels 0.14.6 ----- IPython 9.8.0 jupyter_client 8.7.0 jupyter_core 5.9.1 ----- Python 3.13.11 | packaged by Anaconda, Inc. | (main, Dec 10 2025, 21:28:48) [GCC 14.3.0] Linux-6.14.0-37-generic-x86_64-with-glibc2.39 ----- Session information updated at 2026-01-11 16:02
Bibliografía¶
Linear Models with R by Julian J.Faraway
OpenIntro Statistics: Fourth Edition by David Diez, Mine Çetinkaya-Rundel, Christopher Barr
Introduction to Machine Learning with Python: A Guide for Data Scientists
Krzywinski, M., Altman, N. Multiple linear regression. Nat Methods 12, 1103–1104 (2015)
Instrucciones para citar¶
¿Cómo citar este documento?
Si utilizas este documento o alguna parte de él, te agradecemos que lo cites. ¡Muchas gracias!
Regresión lineal múltiple con Python por Joaquín Amat Rodrigo, disponible con una licencia Attribution-NonCommercial-ShareAlike 4.0 International (CC BY-NC-SA 4.0 DEED) en https://cienciadedatos.net/documentos/py10b-regresion-lineal-multiple-python.html
¿Te ha gustado el artículo? Tu ayuda es importante
Tu contribución me ayudará a seguir generando contenido divulgativo gratuito. ¡Muchísimas gracias! 😊
Este documento creado por Joaquín Amat Rodrigo tiene licencia Attribution-NonCommercial-ShareAlike 4.0 International.
Se permite:
-
Compartir: copiar y redistribuir el material en cualquier medio o formato.
-
Adaptar: remezclar, transformar y crear a partir del material.
Bajo los siguientes términos:
-
Atribución: Debes otorgar el crédito adecuado, proporcionar un enlace a la licencia e indicar si se realizaron cambios. Puedes hacerlo de cualquier manera razonable, pero no de una forma que sugiera que el licenciante te respalda o respalda tu uso.
-
No-Comercial: No puedes utilizar el material para fines comerciales.
-
Compartir-Igual: Si remezclas, transformas o creas a partir del material, debes distribuir tus contribuciones bajo la misma licencia que el original.

