Más sobre ciencia de datos en: cienciadedatos.net
- Regresión lineal con Python
- Regresión lineal múltiple con Python
- Regresión logística con Python
- Regularización Ridge, Lasso y Elastic Net con Python
- Machine learning con Python y Scikitlearn
- Árboles de decisión con Python: regresión y clasificación
- Random Forest con Python y Scikit-learn
- Gradient Boosting con Python y Scikit-learn
- Gradient Boosting probabilístico con Python
- Máquinas de Vector Soporte (SVM)
- Redes neuronales con Python
- Análisis de componentes principales PCA
- Clustering
- Detección de anomalías con PCA
- Detección de anomalías con autoencoders
- Detección de anomalías con Gaussian Mixture Models
- Detección de anomalías con Isolation Forest
- Análisis de texto (text mining) con Python
- Reglas de asociación
- Análisis Tweets con Python

Introducción¶
Los árboles de decisión son modelos predictivos formados por reglas binarias (si/no) con las que se consigue repartir las observaciones en función de sus atributos y predecir así el valor de la variable respuesta.
Muchos métodos predictivos generan modelos globales en los que una única ecuación se aplica a todo el espacio muestral. Cuando el caso de uso implica múltiples predictores, que interaccionan entre ellos de forma compleja y no lineal, es muy difícil encontrar un único modelo global que sea capaz de reflejar la relación entre las variables. Los métodos estadísticos y de machine learning basados en árboles engloban a un conjunto de técnicas supervisadas no paramétricas que consiguen segmentar el espacio de los predictores en regiones simples, dentro de las cuales es más sencillo manejar las interacciones. Es esta característica la que les proporciona gran parte de su potencial.
Los métodos basados en árboles se han convertido en uno de los referentes dentro del ámbito predictivo debido a los buenos resultados que generan en problemas muy diversos. A lo largo de este documento se explora la forma en que se construyen y predicen los árboles de decisión (clasificación y regresión), elementos fundamentales de modelos predictivos más complejos como Random Forest y Gradient Boosting Machine.
Ventajas
Los árboles son fáciles de interpretar aun cuando las relaciones entre predictores son complejas.
Los modelos basados en un solo árbol (no es el caso de random forest, boosting) se pueden representar gráficamente aun cuando el número de predictores es mayor de 3.
Los árboles pueden, en teoría, manejar tanto predictores numéricos como categóricos sin tener que crear variables dummy o one-hot-encoding. En la práctica, esto depende de la implementación del algoritmo que tenga cada librería.
Al tratarse de métodos no paramétricos, no es necesario que se cumpla ningún tipo de distribución específica.
Por lo general, requieren mucha menos limpieza y preprocesado de los datos en comparación con otros métodos de aprendizaje estadístico (por ejemplo, no requieren estandarización).
No se ven muy influenciados por outliers.
Si para alguna observación, el valor de un predictor no está disponible, a pesar de no poder llegar a ningún nodo terminal, se puede conseguir una predicción empleando todas las observaciones que pertenecen al último nodo alcanzado. La precisión de la predicción se verá reducida pero al menos podrá obtenerse.
Son muy útiles en la exploración de datos, permiten identificar de forma rápida y eficiente las variables (predictores) más importantes.
Son capaces de seleccionar predictores de forma automática.
Pueden aplicarse a problemas de regresión y clasificación.
Desventajas
La capacidad predictiva de los modelos basados en un único árbol es bastante inferior a la conseguida con otros modelos. Esto es debido a su tendencia al overfitting y alta varianza. Sin embargo, existen técnicas más complejas que, haciendo uso de la combinación de múltiples árboles (bagging, random forest, boosting), consiguen mejorar en gran medida este problema.
Son sensibles a datos de entrenamiento desbalanceados (una de las clases domina sobre las demás).
Cuando tratan con predictores continuos, pierden parte de su información al categorizarlos en el momento de la división de los nodos.
Tal y como se describe más adelante, la creación de las ramificaciones de los árboles se consigue mediante el algoritmo de recursive binary splitting. Este algoritmo identifica y evalúa las posibles divisiones de cada predictor acorde a una determinada medida (RSS, Gini, entropía…). Los predictores continuos tienen mayor probabilidad de contener, solo por azar, algún punto de corte óptimo, por lo que suelen verse favorecidos en la creación de los árboles.
No son capaces de extrapolar fuera del rango de los predictores observado en los datos de entrenamiento.
Arboles de decisión en Python
La principal implementación de árboles de decisión en Python está disponible en la librería scikit-learn a través de las clases DecisionTreeClassifier y DecisionTreeRegressor. Una característica importante para aquellos que han utilizado otras implementaciones es que, en scikit-learn, es necesario convertir las variables categóricas en variables dummy (one-hot-encoding).
Árboles de regresión¶
Los árboles de regresión son el subtipo de árboles de predicción que se aplica cuando la variable respuesta es continua. En términos generales, en el entrenamiento de un árbol de regresión, las observaciones se van distribuyendo por bifurcaciones (nodos) generando la estructura del árbol hasta alcanzar un nodo terminal. Cuando se quiere predecir una nueva observación, se recorre el árbol acorde al valor de sus predictores hasta alcanzar uno de los nodos terminales. La predicción del árbol es la media de la variable respuesta de las observaciones de entrenamiento que están en ese mismo nodo terminal.
Entrenamiento del árbol¶
El proceso de entrenamiento de un árbol de predicción (regresión o clasificación) se divide en dos etapas:
División sucesiva del espacio de los predictores generando regiones no solapantes (nodos terminales) $R_1$, $R_2$, $R_3$, ..., $R_j$. Aunque, desde el punto de vista teórico las regiones podrían tener cualquier forma, si se limitan a regiones rectangulares (de múltiples dimensiones), se simplifica en gran medida el proceso de construcción y se facilita la interpretación.
Predicción de la variable respuesta en cada región.
A pesar de la sencillez con la que se puede resumir el proceso de construcción de un árbol, es necesario establecer una metodología que permita crear las regiones $R_1$, $R_2$, $R_3$, ..., $R_j$, o lo que es equivalente, decidir donde se introducen las divisiones: en que predictores y en que valores de los mismos. Es en este punto donde se diferencian lo algoritmos de árboles de regresión y clasificación.
En los árboles de regresión, el criterio empleado con más frecuencia para identificar las divisiones es el Residual Sum of Squares (RSS). El objetivo es encontrar las $J$ regiones $(R_1$,..., $R_j)$ que minimizan el Residual Sum of Squares (RSS) total:
$$RSS=\displaystyle\sum_{j=1}^J \displaystyle\sum_{i \in R_j}(y_i -\hat{y}_{R_j})^2 ,$$donde $\hat{y}_{R_j}$ es la media de la variable respuesta en la región $R_j$. Una descripción menos técnica equivale a decir que se busca una distribución de regiones tal que, el sumatorio de las desviaciones al cuadrado entre las observaciones y la media de la región a la que pertenecen sea lo menor posible.
Desafortunadamente, no es posible considerar todas las posibles particiones del espacio de los predictores. Por esta razón, se recurre a lo que se conoce como recursive binary splitting (división binaria recursiva). Esta solución sigue la misma idea que la selección de predictores stepwise (backward o fordward) en regresión lineal múltiple, no evalúa todas las posibles regiones pero, alcanza un buen balance computación-resultado.
Recursive binary splitting
El objetivo del método recursive binary splitting es encontrar, en cada iteración, el predictor $X_j$ y el punto de corte (umbral) $s$ tal que, si se distribuyen las observaciones en las regiones $\{X|X_j < s \}$ y $\{X|X_j \geq s \}$, se consigue la mayor reducción posible en el RSS. El algoritmo seguido es:
El proceso se inicia en lo más alto del árbol, donde todas las observaciones pertenecen a la misma región.
Se identifican todos los posibles puntos de corte $s$ para cada uno de los predictores ($X_1$, $X_2$,..., $X_p$). En el caso de predictores cualitativos, los posibles puntos de corte son cada uno de sus niveles. Para predictores continuos, se ordenan de menor a mayor sus valores, el punto intermedio entre cada par de valores se emplea como punto de corte.
Se calcula el RSS total que se consigue con cada posible división identificada en el paso 2.
donde el primer término es el RSS de la región 1 y el segundo término es el RSS de la región 2, siendo cada una de las regiones el resultado de separar las observaciones acorde al predictor $j$ y valor $s$.
Se selecciona el predictor $X_j$ y el punto de corte $S$ que resulta en el menor RSS total, es decir, que da lugar a las divisiones más homogéneas posibles. Si existen dos o más divisiones que consiguen la misma mejora, la elección entre ellas es aleatoria.
Se repiten de forma iterativa los pasos 1 a 4 para cada una de las regiones que se han creado en la iteración anterior hasta que se alcanza alguna norma de stop. Algunas de las más empleadas son: alcanzar una profundidad máxima, que ninguna región contenga menos de n observaciones, que el árbol tenga un máximo de nodos terminales o que la incorporación del más nodos no reduzca el error en al menos un % mínimo.
Está metodología conlleva dos hechos:
Que cada división óptima se identifica acorde al impacto que tiene en ese momento. No se tiene en cuenta si es la división que dará lugar a mejores árboles en futuras divisiones.
En cada división se evalúa un único predictor haciendo preguntas binarias (si, no), lo que genera dos nuevas ramas del árbol por división. A pesar de que es posible evaluar divisiones más complejas, hacer una pregunta sobre múltiples variables a la vez es equivalente a hacer múltiples preguntas sobre variables individuales.
✏️ Nota
Los algoritmos que implementan recursive binary splitting suelen incorporar estrategias para evitar evaluar todos los posibles puntos de corte. Por ejemplo, para predictores continuos, primero se crea un histograma que agrupa los valores y luego se evaluan los puntos de corte de cada región del histograma (bin).
Predicción del árbol¶
Tras la creación de un árbol, las observaciones de entrenamiento quedan agrupadas en los nodos terminales. Para predecir una nueva observación, se recorre el árbol en función de los valores que tienen sus predictores hasta llegar a uno de los nodos terminales. En el caso de regresión, el valor predicho suele ser la media de la variable respuesta de las observaciones de entrenamiento que están en ese mismo nodo. Si bien la media es valor más empleado, se puede utilizar cualquier otro (mediana, cuantil...).
Supóngase que se dispone de 10 observaciones, cada una con un valor de variable respuesta $Y$ y unos predictores $X$.
| id | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 |
| Y | 10 | 18 | 24 | 8 | 2 | 9 | 16 | 10 | 20 | 14 |
| X | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
La siguiente imagen muestra cómo sería la predicción del árbol para una nueva observación. El camino hasta llegar al nodo final está resaltado. En cada nodo terminal se detalla el índice de las observaciones de entrenamiento que forman parte.
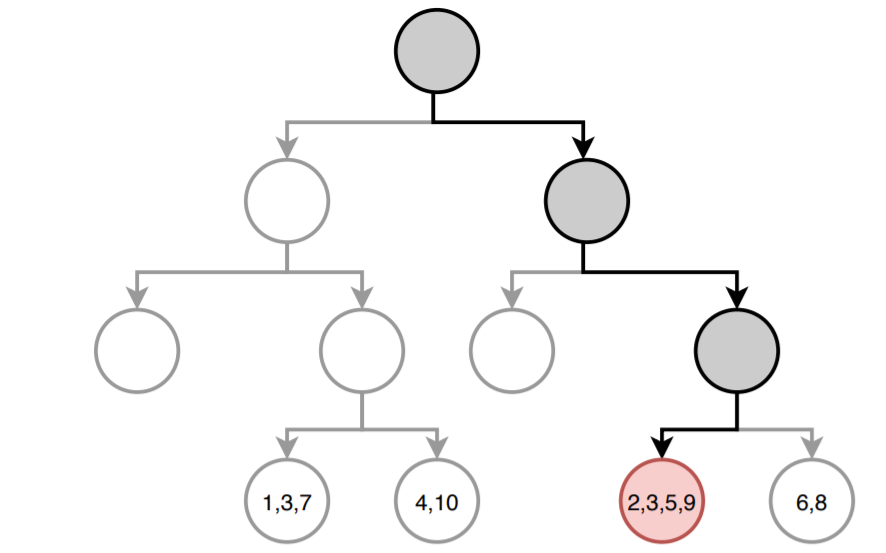
El valor predicho por el árbol es la media de la variable respuesta $Y$ de las observaciones con $id$: 2, 3, 5, 9.
$$\hat{\mu} = \frac{18+24+2+20}{4} = 16$$Aunque la anterior es la forma más común de obtener las predicciones de un árbol de regresión, existe otra aproximación. La predicción de un árbol de regresión puede verse como una variante de vecinos cercanos en la que, solo las observaciones que forman parte del mismo nodo terminal que la observación predicha, tienen influencia. Siguiendo esta aproximación, la predicción del árbol se define como la media ponderada de todas las observaciones de entrenamiento, donde el peso de cada observación depende únicamente de si forma parte o no del mismo nodo terminal.
$$\hat{\mu} = \sum^n_{i =1} \textbf{w}_iY_i$$El valor de las posiciones del vector de pesos $\textbf{w}$ es $1$ para las observaciones que están en el mismo nodo y $0$ para el resto. En este ejemplo sería:
$$\textbf{w} =(0, 1, 1, 0, 1, 0, 0, 0, 1, 0)$$Para que la suma de todos los pesos sea $1$, se dividen por el número total de observaciones en el nodo terminal seleccionado, en este caso 4.
$$\textbf{w} = (0, \frac{1}{4}, \frac{1}{4}, 0, \frac{1}{4}, 0, 0, 0, \frac{1}{4}, 0)$$Así pues, el valor predicho es:
$$\hat{\mu} = (0\times10) + (\frac{1}{4}\times18) + (\frac{1}{4}\times24) + (0\times8) + (\frac{1}{4}\times2) + \\ (0\times9) + (0\times16) + (0\times10) + (\frac{1}{4}\times20) + (0\times14) = 16$$Árboles de clasificación¶
Los árboles de regresión son el subtipo de árboles de predicción que se aplica cuando la variable respuesta es categórica.
Construcción del árbol¶
Para construir un árbol de clasificación, se emplea el mismo método recursive binary splitting descrito en los árboles de regresión. Sin embargo, como la variable respuesta en cualitativa, no se puede emplear el RSS como criterio de selección de las divisiones óptimas. Existen varias alternativas, todas ellas con el objetivo de encontrar nodos lo más puros/homogéneos posible. Las más empleadas son:
CLASSIFICATION ERROR RATE
Se define como la proporción de observaciones que no pertenecen a la clase mayoritaria del nodo.
$$E_m = 1 - max_k(\hat{p}_{mk})$$donde $\hat{p}_{mk}$ representa la proporción de observaciones del nodo $m$ que pertenecen a la clase $k$. A pesar de la sencillez de esta medida, no es suficientemente sensible para crear buenos árboles, por lo que, en la práctica, no suele emplearse.
GINI INDEX
Cuantifica la varianza total en el conjunto de las $K$ clases del nodo $m$, es decir, mide la pureza del nodo.
$$G_m = \sum_{k=1}^K \hat{p}_{mk}(1-\hat{p}_{mk})$$Cuando $\hat{p}_{mk}$ es cercano a 0 o a 1 (el nodo contiene mayoritariamente observaciones de una sola clase), el término $\hat{p}_{mk}(1-\hat{p}_{mk})$ es muy pequeño. Como consecuencia, cuanto mayor sea la pureza del nodo, menor el valor del índice Gini $G$.
El algoritmo CART (Classification and Regression Trees) emplea Gini como criterio de división.
INFORMATION GAIN: CROSS ENTROPY
La entropía es una forma de cuantificar el desorden de un sistema. En el caso de los nodos, el desorden se corresponde con la impureza. Si un nodo es puro, contiene únicamente observaciones de una clase, su entropía es cero. Por el contrario, si la frecuencia de cada clase es la misma, el valor de la entropía alcanza el valor máximo de 1.
$$D = - \displaystyle\sum_{k=1}^K \hat{p}_{mk} \ log(\hat{p}_{mk})$$Los algoritmos C4.5 y C5.0 emplean information gain como criterio de división.
CHI-SQUARE
Esta aproximación consiste en identificar si existe una diferencia significativa entre los nodos hijos y el nodo parental, es decir, si hay evidencias de que la división consigue una mejora. Para ello, se aplica un test estadístico chi-square goodness of fit empleando como distribución esperada $H_0$ la frecuencia de cada clase en el nodo parental. Cuanto mayor el estadístico $\chi^2$, mayor la evidencia estadística de que existe una diferencia.
$$\chi^2 = \sum_{k} \frac{(\text{observado}_{k} - \text{esperado}_{k})^2}{\text{esperado}_{k}}$$Los árboles generados con este criterio de división reciben el nombre de CHAID (Chi-square Automatic Interaction Detector).
Independientemente de la medida empleada como criterio de selección de las divisiones, el proceso siempre es el mismo:
Para cada posible división se calcula el valor de la medida en cada uno de los dos nodos resultantes.
Se suman los dos valores, ponderando cada uno por la fracción de observaciones que contiene cada nodo. Este paso es muy importante, ya que no es lo mismo dos nodos puros con 2 observaciones, que dos nodos puros con 100 observaciones.
- La división con menor o mayor valor (dependiendo de la medida empleada) se selecciona como punto de corte óptimo.
Para el proceso de construcción del árbol, acorde al libro Introduction to Statistical Learning, Gini index y cross-entropy son más adecuados que el classification error rate debido a su mayor sensibilidad a la homogeneidad de los nodos. Para el proceso de pruning (descrito en la siguiente sección) los tres son adecuados, aunque, si el objetivo es conseguir la máxima precisión en las predicciones, mejor emplear el classification error rate.
Predicción del árbol¶
Tras la creación de un árbol, las observaciones de entrenamiento quedan agrupadas en los nodos terminales. Para predecir una nueva observación, se recorre el árbol en función del valor de sus predictores hasta llegar a uno de los nodos terminales. En el caso de clasificación, suele emplearse la moda de la variable respuesta como valor de predicción, es decir, la clase más frecuente del nodo. Además, puede acompañarse con el porcentaje de cada clase en el nodo terminal, lo que aporta información sobre la confianza de la predicción.
Evitar el overfitting¶
El proceso de construcción de árboles descrito en las secciones anteriores tiende a reducir rápidamente el error de entrenamiento, es decir, el modelo se ajusta muy bien a las observaciones empleadas como entrenamiento. Como consecuencia, se genera un overfitting que reduce su capacidad predictiva al aplicarlo a nuevos datos. La razón de este comportamiento radica en la facilidad con la que los árboles se ramifican adquiriendo estructuras complejas. De hecho, si no se limitan las divisiones, todo árbol termina ajustándose perfectamente a las observaciones de entrenamiento creando un nodo terminal por observación. Existen dos estrategias para prevenir el problema de overfitting de los árboles: limitar el tamaño del árbol (parada temprana) y el proceso de podado (pruning).
Parada temprana (early stopping)¶
El tamaño final que adquiere un árbol puede controlarse mediante reglas de parada que detengan la división de los nodos dependiendo de si se cumplen o no determinadas condiciones. El nombre de estas condiciones puede variar dependiendo del software o librería empleada, pero suelen estar presentes en todos ellos.
Observaciones mínimas para división: define el número mínimo de observaciones que debe tener un nodo para poder ser dividido. Cuanto mayor el valor, menos flexible es el modelo.
Observaciones mínimas de nodo terminal: define el número mínimo de observaciones que deben tener los nodos terminales. Su efecto es muy similar al de observaciones mínimas para división.
Profundidad máxima del árbol: define la profundidad máxima del árbol, entendiendo por profundidad máxima el número de divisiones de la rama más larga (en sentido descendente) del árbol. Cuanto menor el valor, menos flexible es el modelo.
Número máximo de nodos terminales: define el número máximo de nodos terminales que puede tener el árbol. Una vez alcanzado el límite, se detienen las divisiones. Su efecto es similar al de controlar la profundidad máxima del árbol.
Reducción mínima de error: define la reducción mínima de error que tiene que conseguir una división para que se lleve a cabo.
Todos estos parámetros son lo que se conoce como hiperparámetros porque no se aprenden durante el entrenamiento del modelo. Su valor tiene que ser especificado por el usuario en base a su conocimiento del problema y mediante el uso de estrategias de validación.
Tree pruning¶
La estrategia de controlar el tamaño del árbol mediante reglas de parada tiene un inconveniente, el árbol se crece seleccionando la mejor división en cada momento. Al evaluar las divisiones sin tener en cuenta las que vendrán después, nunca se elige la opción que resulta en el mejor árbol final, a no ser que también sea la que genera en ese momento la mejor división. A este tipo de estrategias se les conoce como greedy. Un ejemplo que ilustra el problema de este tipo de estrategia es el siguiente: supóngase que un coche circula por el carril izquierdo de una carretera de dos carriles en la misma dirección. En el carril que se encuentra hay muchos coches circulando a 100 km/h, mientras que el otro carril se encuentra vacío. A cierta distancia se observa que hay un vehículo circulando por el carril derecho a 20 km/h. Si el objetivo del conductor es llegar a su destino lo antes posible tiene dos opciones: cambiarse de carril o mantenerse en el que está. Una aproximación de tipo greedy evaluaría la situación en ese instante y determinaría que la mejor opción es cambiarse de carril y acelerar a más de 100 km/h, sin embargo, a largo plazo, esta no es la mejor solución, ya que una vez alcance al vehículo lento, tendrá que reducir mucho su velocidad.
Una alternativa no greedy que consigue evitar el overfitting consiste en generar árboles grandes, sin condiciones de parada más allá de las necesarias por las limitaciones computacionales, y después podarlos (pruning), manteniendo únicamente la estructura robusta que consigue un test error bajo. La selección del sub-árbol óptimo puede hacerse mediante cross-validation, sin embargo, dado que los árboles se crecen lo máximo posible (tienen muchos nodos terminales) no suele ser viable estimar el test error de todas las posibles sub-estructuras que se pueden generar. En su lugar, se recurre al cost complexity pruning o weakest link pruning.
Cost complexity pruning es un método de penalización de tipo Loss + Penalty, similar al empleado en ridge regression o lasso. En este caso, se busca el sub-árbol $T$ que minimiza la ecuación:
$$\displaystyle\sum_{j=1}^{|T|} \displaystyle\sum_{i \in R_j}(y_i -\hat{y}_{R_j})^2 + \alpha|T| $$donde $|T|$ es el número de nodos terminales del árbol.
El primer término de la ecuación se corresponde con el sumatorio total de los residuos cuadrados RSS. Por definición, cuantos más nodos terminales tenga el modelo, menor será esta parte de la ecuación. El segundo término es la restricción, que penaliza al modelo en función del número de nodos terminales (a mayor número, mayor penalización). El grado de penalización se determina mediante el tuning parameter $\alpha$. Cuando $\alpha = 0$, la penalización es nula y el árbol resultante es equivalente al árbol original. A medida que se incrementa $\alpha$, la penalización es mayor y, como consecuencia, los árboles resultantes son de menor tamaño. El valor óptimo de $\alpha$ puede identificarse mediante cross validation.
Algoritmo para crear un árbol de regresión con pruning
Se emplea recursive binary splitting para crear un árbol grande y complejo ($T_o$) empleando los datos de entrenamiento y reduciendo al máximo posible las condiciones de parada. Normalmente se emplea como única condición de parada el número mínimo de observaciones por nodo terminal.
Se aplica el cost complexity pruning al árbol $T_o$ para obtener el mejor sub-árbol en función de $\alpha$. Es decir, se obtiene el mejor sub-árbol para un rango de valores de $\alpha$.
Identificación del valor óptimo de $\alpha$ mediante k-cross-validation. Se divide el *conjunto de entrenamiento en $K$ grupos. Para $k=1$, ..., $k=K$:
Repetir pasos 1 y 2 empleando todas las observaciones excepto las del grupo $k_i$.
Evaluar el mean squared error para el rango de valores de $\alpha$ empleando el grupo $k_i$.
Obtener el promedio de los $K$ mean squared error calculados para cada valor $\alpha$.
Seleccionar el sub-árbol del paso 2 que se corresponde con el valor $\alpha$ que ha conseguido el menor cross-validation mean squared error en el paso 3.
En el caso de los árboles de clasificación, en lugar de emplear la suma de residuos cuadrados como criterio de selección, se emplea alguna de las medidas de homogeneidad vistas en apartados anteriores.
Ejemplo regresión¶
Librerías¶
Las librerías utilizadas en este ejemplo son:
# Tratamiento de datos
# ==============================================================================
import numpy as np
import pandas as pd
# Gráficos
# ==============================================================================
import matplotlib.pyplot as plt
# Preprocesado y modelado
# ==============================================================================
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeRegressor
from sklearn.tree import plot_tree
from sklearn.tree import export_text
from sklearn.model_selection import GridSearchCV
from sklearn.metrics import root_mean_squared_error
# Configuración warnings
# ==============================================================================
import warnings
warnings.filterwarnings('once')
Datos¶
El set de datos Boston contiene precios de viviendas de la ciudad de Boston, así como información socio-económica del barrio en el que se encuentran. Se pretende ajustar un modelo de regresión que permita predecir el precio medio de una vivienda (MEDV) en función de las variables disponibles.
Número de instancias: 506
Número de atributos: 13 predictivos numéricos/categoriales.
Información de los atributos (por orden):
- CRIM: tasa de delincuencia per cápita por ciudad.
- ZN: proporción de suelo residencial dividido para segmentos de más de 25.000 pies cuadrados.
- INDUS: proporción de superficie comerciales no minoristas por ciudad.
- CHAS: variable binaria sobre el río Charles (= 1 si la zona linda con el río; 0 en caso contrario).
- NOX: concentración de óxidos nítricos (partes por 10 millones).
- RM: número medio de habitaciones por vivienda.
- EDAD: proporción de viviendas ocupadas construidas antes de 1940.
- DIS: distancia ponderada a cinco centros de empleo en la ciudad de Boston.
- RAD: índice de accesibilidad a las autopistas radiales.
- TAX: impuesto sobre bienes inmuebles de valor íntegro por cada 10.000$.
- PTRATIO: ratio alumnos-profesor por ciudad.
- LSTAT: % de la población considerado de clase baja.
- MEDV: valor medio de las viviendas ocupadas por sus propietarios en miles de $.
Valores ausentes: Ninguno
# Descarga de datos
# ==============================================================================
url = (
'https://raw.githubusercontent.com/JoaquinAmatRodrigo/Estadistica-machine-learning-python/'
'master/data/Boston.csv'
)
datos = pd.read_csv(url, sep=',')
display(datos.head(3))
display(datos.info())
| CRIM | ZN | INDUS | CHAS | NOX | RM | AGE | DIS | RAD | TAX | PTRATIO | LSTAT | MEDV | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0.00632 | 18.0 | 2.31 | 0 | 0.538 | 6.575 | 65.2 | 4.0900 | 1 | 296 | 15.3 | 4.98 | 24.0 |
| 1 | 0.02731 | 0.0 | 7.07 | 0 | 0.469 | 6.421 | 78.9 | 4.9671 | 2 | 242 | 17.8 | 9.14 | 21.6 |
| 2 | 0.02729 | 0.0 | 7.07 | 0 | 0.469 | 7.185 | 61.1 | 4.9671 | 2 | 242 | 17.8 | 4.03 | 34.7 |
<class 'pandas.core.frame.DataFrame'> RangeIndex: 506 entries, 0 to 505 Data columns (total 13 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 CRIM 506 non-null float64 1 ZN 506 non-null float64 2 INDUS 506 non-null float64 3 CHAS 506 non-null int64 4 NOX 506 non-null float64 5 RM 506 non-null float64 6 AGE 506 non-null float64 7 DIS 506 non-null float64 8 RAD 506 non-null int64 9 TAX 506 non-null int64 10 PTRATIO 506 non-null float64 11 LSTAT 506 non-null float64 12 MEDV 506 non-null float64 dtypes: float64(10), int64(3) memory usage: 51.5 KB
None
Ajuste del modelo¶
La clase DecisionTreeRegressor del módulo sklearn.tree permite entrenar árboles de decisión para problemas de regresión. A continuación, se ajusta un árbol de regresión empleando como variable respuesta MEDV y como predictores todas las otras variables disponibles.
DecisionTreeRegressor tiene por defecto los siguientes hiperparámetros:
criterion='squared_error'splitter='best'max_depth=Nonemin_samples_split=2min_samples_leaf=1min_weight_fraction_leaf=0.0max_features=Nonerandom_state=Nonemax_leaf_nodes=Nonemin_impurity_decrease=0.0min_impurity_split=Noneccp_alpha=0.0monotonic_cst
De entre todos ellos, los más importantes son aquellos que detienen el crecimiento del árbol (condiciones de stop):
max_depth: profundidad máxima que puede alcanzar el árbol.min_samples_split: número mínimo de observaciones que debe de tener un nodo para que pueda dividirse. Si es un valor decimal se interpreta como fracción del total de observaciones de entrenamientoceil(min_samples_split * n_samples).min_samples_leaf: número mínimo de observaciones que debe de tener cada uno de los nodos hijos para que se produzca la división. Si es un valor decimal se interpreta como fracción del total de observaciones de entrenamientoceil(min_samples_split * n_samples).max_leaf_nodes: número máximo de nodos terminales.random_state: semilla para que los resultados sean reproducibles. Tiene que ser un valor entero.
Como en todo estudio de regresión, no solo es importante ajustar el modelo, sino también cuantificar su capacidad para predecir nuevas observaciones. Para poder hacer la posterior evaluación, se dividen los datos en dos grupos, uno de entrenamiento y otro de test.
# División de los datos en train y test
# ==============================================================================
X_train, X_test, y_train, y_test = train_test_split(
datos.drop(columns = "MEDV"),
datos['MEDV'],
random_state = 123
)
# Creación del modelo
# ==============================================================================
modelo = DecisionTreeRegressor(max_depth=3, random_state=123)
# Entrenamiento del modelo
# ==============================================================================
modelo.fit(X_train, y_train)
DecisionTreeRegressor(max_depth=3, random_state=123)In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
Parameters
| criterion | 'squared_error' | |
| splitter | 'best' | |
| max_depth | 3 | |
| min_samples_split | 2 | |
| min_samples_leaf | 1 | |
| min_weight_fraction_leaf | 0.0 | |
| max_features | None | |
| random_state | 123 | |
| max_leaf_nodes | None | |
| min_impurity_decrease | 0.0 | |
| ccp_alpha | 0.0 | |
| monotonic_cst | None |
Una vez entrenado el árbol, se puede representar mediante la combinación de las funciones plot_tree() y export_text(). La función plot_tree() dibuja la estructura del árbol y muestra el número de observaciones y valor medio de la variable respuesta en cada nodo. La función export_text() representa esta misma información en formato texto
# Estructura del árbol creado
# ==============================================================================
fig, ax = plt.subplots(figsize=(9, 5))
print(f"Profundidad del árbol: {modelo.get_depth()}")
print(f"Número de nodos terminales: {modelo.get_n_leaves()}")
plot = plot_tree(
decision_tree = modelo,
feature_names = datos.drop(columns = "MEDV").columns,
class_names = ['MEDV'],
filled = True,
impurity = False,
fontsize = 8,
precision = 2,
ax = ax
)
Profundidad del árbol: 3 Número de nodos terminales: 8
# Estructura en formato texto
# ==============================================================================
texto_modelo = export_text(
decision_tree = modelo,
feature_names = list(datos.drop(columns = "MEDV").columns)
)
print(texto_modelo)
|--- RM <= 6.98 | |--- LSTAT <= 14.39 | | |--- DIS <= 1.42 | | | |--- value: [50.00] | | |--- DIS > 1.42 | | | |--- value: [23.13] | |--- LSTAT > 14.39 | | |--- CRIM <= 6.99 | | | |--- value: [16.96] | | |--- CRIM > 6.99 | | | |--- value: [11.58] |--- RM > 6.98 | |--- RM <= 7.44 | | |--- DIS <= 1.57 | | | |--- value: [50.00] | | |--- DIS > 1.57 | | | |--- value: [32.17] | |--- RM > 7.44 | | |--- PTRATIO <= 17.90 | | | |--- value: [47.36] | | |--- PTRATIO > 17.90 | | | |--- value: [40.12]
Siguiendo la rama más a la izquierda del árbol, puede verse que el modelo predice un precio promedio de 50000 dólares para viviendas que están en una zona con un RM <= 6.98, un LSTAT <= 14.39 y un DIS <= 1.42.
Importancia de predictores¶
La importancia de cada predictor en modelo se calcula como la reducción total (normalizada) en el criterio de división, en este caso el mse, que consigue el predictor en las divisiones en las que participa. Si un predictor no ha sido seleccionado en ninguna divisón, no se ha incluido en el modelo y por lo tanto su importancia es 0.
importancia_predictores = pd.DataFrame(
{
"predictor": datos.drop(columns="MEDV").columns,
"importancia": modelo.feature_importances_,
}
)
print("Importancia de los predictores en el modelo")
print("-------------------------------------------")
importancia_predictores.sort_values("importancia", ascending=False)
Importancia de los predictores en el modelo -------------------------------------------
| predictor | importancia | |
|---|---|---|
| 5 | RM | 0.671680 |
| 11 | LSTAT | 0.222326 |
| 7 | DIS | 0.064816 |
| 0 | CRIM | 0.034714 |
| 10 | PTRATIO | 0.006465 |
| 3 | CHAS | 0.000000 |
| 1 | ZN | 0.000000 |
| 2 | INDUS | 0.000000 |
| 6 | AGE | 0.000000 |
| 4 | NOX | 0.000000 |
| 9 | TAX | 0.000000 |
| 8 | RAD | 0.000000 |
El predictor RM, el número medio de habitaciones en las viviendas de la zona, ha resultado ser el predictor más importante en el modelo, seguido de LSTAT, que mide el porcentaje de personas en estado de pobreza.
Podado del árbol (pruning)¶
Con la finalidad de reducir la varianza del modelo y así mejorar la capacidad predictiva, se somete al árbol a un proceso de pruning. Tal como se describió con anterioridad, el proceso de pruning intenta encontrar el árbol más sencillo (menor tamaño) que consigue los mejores resultados de predicción.
Para aplicar el proceso de pruning es necesario indicar el argumento ccp_alpha que determina el grado de penalización por complejidad. Cuanto mayor es este valor, más agresivo el podado y menor el tamaño del árbol resultante. Dado que no hay forma de conocer de antemano el valor óptimo de ccp_alpha, se recurre a validación cruzada para identificarlo.
Aunque existen otras formas de indentificar árboles "optimos", por ejemplo identificando el valor de max_depth y min_samples_split mediante validación cruzada, el pruning puede generar mejores resultados ya que permite que una división poco útil se mantenga si las siguientes divisiones que parten de ella sí lo son.
# Pruning (const complexity pruning) por validación cruzada
# ==============================================================================
# Valores de ccp_alpha evaluados
param_grid = {'ccp_alpha': np.linspace(0, 20, 20)}
# Búsqueda por validación cruzada
grid = GridSearchCV(
# El árbol se crece al máximo posible para luego aplicar el pruning
estimator = DecisionTreeRegressor(
max_depth = None,
min_samples_split = 2,
min_samples_leaf = 1,
random_state = 123
),
param_grid = param_grid,
scoring = 'neg_mean_squared_error',
cv = 10,
refit = True,
return_train_score = True
)
grid.fit(X_train, y_train)
fig, ax = plt.subplots(figsize=(8, 3.84))
scores = pd.DataFrame(grid.cv_results_)
# Convertimos a error positivo para facilitar la lectura (MSE)
scores['mean_train_score'] = -scores['mean_train_score']
scores['mean_test_score'] = -scores['mean_test_score']
scores.plot(x='param_ccp_alpha', y='mean_train_score', yerr='std_train_score', ax=ax, marker='o', label='Train Error')
scores.plot(x='param_ccp_alpha', y='mean_test_score', yerr='std_test_score', ax=ax, marker='o', label='CV Error')
# Marcamos el mejor alpha
best_alpha = grid.best_params_['ccp_alpha']
ax.axvline(x=best_alpha, color='red', linestyle='--', label=f'Mejor alpha: {best_alpha:.2f}')
ax.set_title("Error de validación cruzada (MSE) vs hiperparámetro ccp_alpha")
ax.set_ylabel("Mean Squared Error")
ax.legend();
# Mejor valor ccp_alpha encontrado
# ==============================================================================
grid.best_params_
{'ccp_alpha': np.float64(1.0526315789473684)}
Una vez identificado el valor óptimo de ccp_alpha, se reentrena el árbol indicando este valor en sus argumentos. Si en el GridSearchCV() se indica refit=True, este reentrenamiento se hace automáticamente y
el modelo resultante se encuentra almacenado en .best_estimator_.
# Estructura del árbol final
# ==============================================================================
modelo_final = grid.best_estimator_
print(f"Profundidad del árbol: {modelo_final.get_depth()}")
print(f"Número de nodos terminales: {modelo_final.get_n_leaves()}")
fig, ax = plt.subplots(figsize=(7, 5))
plot = plot_tree(
decision_tree = modelo_final,
feature_names = datos.drop(columns = "MEDV").columns,
class_names = ['MEDV'],
filled = True,
impurity = False,
ax = ax
)
Profundidad del árbol: 4 Número de nodos terminales: 7
Predicción y evaluación del modelo¶
Por último, se evalúa la capacidad predictiva del árbol final empleando el conjunto de test.
# Error de test del modelo final (tras aplicar pruning)
# ==============================================================================
predicciones = modelo_final.predict(X = X_test)
rmse = root_mean_squared_error(
y_true = y_test,
y_pred = predicciones,
)
print(f"El error (rmse) de test es: {rmse}")
El error (rmse) de test es: 5.522220519181609
El proceso de pruning ha generado un arbol con una produndidad de 4 y con 7 nodos terminales. Las predicciones del modelo final se alejan en promedio 5.52 unidades (5520 dólares) del valor real.
Ejemplo clasificación¶
Librerías¶
Las librerías utilizadas en este ejemplo son:
# Tratamiento de datos
# ==============================================================================
import numpy as np
import pandas as pd
import statsmodels.api as sm
# Gráficos
# ==============================================================================
import matplotlib.pyplot as plt
# Preprocesado y modelado
# ==============================================================================
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier
from sklearn.tree import plot_tree
from sklearn.tree import export_text
from sklearn.model_selection import GridSearchCV
from sklearn.compose import ColumnTransformer
from sklearn.preprocessing import OneHotEncoder
from sklearn.metrics import (
accuracy_score,
classification_report,
confusion_matrix,
ConfusionMatrixDisplay,
)
# Configuración warnings
# ==============================================================================
import warnings
warnings.filterwarnings("ignore")
Datos¶
El set de datos Carseats, original del paquete de R ISLR y accesible en Python a través de statsmodels.datasets.get_rdataset, contiene información sobre la venta de sillas infantiles en 400 tiendas distintas. Para cada una de las 400 tiendas se han registrado 11 variables. Se pretende generar un modelo de clasificación que permita predecir si una tienda tiene ventas altas (Sales > 8) o bajas (Sales <= 8) en función de todas las variables disponibles.
💡 Tip
Puedes encontrar un listado de todos los set de datos disponibles en Rdatasets
# Decarga de datos
# ==============================================================================
carseats = sm.datasets.get_rdataset("Carseats", "ISLR")
datos = carseats.data
# print(carseats.__doc__)
datos.head(3)
| Sales | CompPrice | Income | Advertising | Population | Price | ShelveLoc | Age | Education | Urban | US | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 9.50 | 138 | 73 | 11 | 276 | 120 | Bad | 42 | 17 | Yes | Yes |
| 1 | 11.22 | 111 | 48 | 16 | 260 | 83 | Good | 65 | 10 | Yes | Yes |
| 2 | 10.06 | 113 | 35 | 10 | 269 | 80 | Medium | 59 | 12 | Yes | Yes |
Como Sales es una variable continua y el objetivo del estudio es clasificar las tiendas según si venden mucho o poco, se crea una nueva variable dicotómica (0, 1) llamada ventas_altas.
datos['ventas_altas'] = np.where(datos.Sales > 8, 1, 0)
# Una vez creada la nueva variable respuesta se descarta la original
datos = datos.drop(columns = 'Sales')
datos.head(3)
| CompPrice | Income | Advertising | Population | Price | ShelveLoc | Age | Education | Urban | US | ventas_altas | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 138 | 73 | 11 | 276 | 120 | Bad | 42 | 17 | Yes | Yes | 1 |
| 1 | 111 | 48 | 16 | 260 | 83 | Good | 65 | 10 | Yes | Yes | 1 |
| 2 | 113 | 35 | 10 | 269 | 80 | Medium | 59 | 12 | Yes | Yes | 1 |
Ajuste del modelo¶
Se ajusta un árbol de clasificación empleando como variable respuesta ventas_altas y como predictores todas las variables disponibles. Se utilizan en primer lugar los hiperparámetros max_depth=5 y criterion='gini', el resto se dejan por defecto. Después, se aplica el proceso de pruning y se comparan los resultados frente al modelo inicial.
A diferencia del ejemplo anterior, en estos datos hay variables categóricas por lo que, antes de entrenar el modelo, es necesario aplicar one-hot-encoding. Puede encontrarse una descripción más detallada de este proceso en Machine learning con Python y Scikit-learn.
# División de los datos en train y test
# ==============================================================================
X_train, X_test, y_train, y_test = train_test_split(
datos.drop(columns = 'ventas_altas'),
datos['ventas_altas'],
random_state = 123
)
# One-hot-encoding de las variables categóricas
# ==============================================================================
# Se identifica el nobre de las columnas numéricas y categóricas
cat_cols = X_train.select_dtypes(include=['object', 'category']).columns.to_list()
numeric_cols = X_train.select_dtypes(include=['float64', 'int']).columns.to_list()
# Se aplica one-hot-encoding solo a las columnas categóricas
preprocessor = ColumnTransformer(
[('onehot', OneHotEncoder(handle_unknown='ignore', sparse_output=False, dtype=float), cat_cols)],
remainder='passthrough',
verbose_feature_names_out=False
).set_output(transform='pandas')
# Una vez que se ha definido el objeto ColumnTransformer, con el método fit()
# se aprenden las transformaciones con los datos de entrenamiento y se aplican a
# los dos conjuntos con transform(). Ambas operaciones a la vez con fit_transform().
X_train_prep = preprocessor.fit_transform(X_train)
X_test_prep = preprocessor.transform(X_test)
X_train_prep.info()
<class 'pandas.core.frame.DataFrame'> Index: 300 entries, 170 to 365 Data columns (total 14 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 ShelveLoc_Bad 300 non-null float64 1 ShelveLoc_Good 300 non-null float64 2 ShelveLoc_Medium 300 non-null float64 3 Urban_No 300 non-null float64 4 Urban_Yes 300 non-null float64 5 US_No 300 non-null float64 6 US_Yes 300 non-null float64 7 CompPrice 300 non-null int64 8 Income 300 non-null int64 9 Advertising 300 non-null int64 10 Population 300 non-null int64 11 Price 300 non-null int64 12 Age 300 non-null int64 13 Education 300 non-null int64 dtypes: float64(7), int64(7) memory usage: 35.2 KB
# Creación del modelo
# ==============================================================================
modelo = DecisionTreeClassifier(
max_depth = 5,
criterion = 'gini',
random_state = 123
)
# Entrenamiento del modelo
# ==============================================================================
modelo.fit(X_train_prep, y_train)
DecisionTreeClassifier(max_depth=5, random_state=123)In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
Parameters
| criterion | 'gini' | |
| splitter | 'best' | |
| max_depth | 5 | |
| min_samples_split | 2 | |
| min_samples_leaf | 1 | |
| min_weight_fraction_leaf | 0.0 | |
| max_features | None | |
| random_state | 123 | |
| max_leaf_nodes | None | |
| min_impurity_decrease | 0.0 | |
| class_weight | None | |
| ccp_alpha | 0.0 | |
| monotonic_cst | None |
# Estructura del árbol creado
# ==============================================================================
fig, ax = plt.subplots(figsize=(10, 6))
print(f"Profundidad del árbol: {modelo.get_depth()}")
print(f"Número de nodos terminales: {modelo.get_n_leaves()}")
plot = plot_tree(
decision_tree = modelo,
feature_names = X_train_prep.columns.to_list(),
class_names = y_train.unique().astype(str).tolist(),
filled = True,
impurity = False,
fontsize = 6,
ax = ax
)
Profundidad del árbol: 5 Número de nodos terminales: 23
Predicción y evaluación del modelo¶
Se evalúa la capacidad predictiva del árbol inicial calculando el accuracy en el conjunto de test.
# Predicciones sobre el conjunto de test
# ==============================================================================
predicciones = modelo.predict(X=X_test_prep)
print(predicciones)
[1 0 1 1 1 0 0 1 0 0 0 0 0 1 0 0 1 0 0 0 0 0 1 1 0 0 1 0 0 0 0 0 0 1 1 0 0 0 0 1 0 0 0 1 0 1 1 0 0 0 0 0 0 1 0 1 1 0 0 1 0 0 0 0 1 1 1 0 0 1 0 0 0 0 1 0 0 0 0 0 1 0 0 0 0 1 0 1 0 0 1 0 1 0 1 1 0 0 1 0]
# Error de test del modelo
# ==============================================================================
print("Matriz de confusión")
print("-------------------")
cm = confusion_matrix(y_true=y_test, y_pred=predicciones)
disp = ConfusionMatrixDisplay(confusion_matrix=cm, display_labels=modelo.classes_)
disp.plot();
Matriz de confusión -------------------
# Informe de clasificación
# ==============================================================================
print(classification_report(y_true=y_test, y_pred=predicciones))
precision recall f1-score support
0 0.66 0.88 0.75 50
1 0.82 0.54 0.65 50
accuracy 0.71 100
macro avg 0.74 0.71 0.70 100
weighted avg 0.74 0.71 0.70 100
El modelo inicial es capaz de predecir correctamente un 71 % de las observaciones del conjunto de test.
Podado del árbol (pruning)¶
Aunque inicialmente se ha empleado un valor de max_depth=5, este no tiene por qué ser el mejor valor. Con el objetivo de identificar la profundidad óptima que consigue reducir la varianza y aumentar la capacidad predictiva del modelo, se somete al árbol a un proceso de pruning.
# Post pruning (const complexity pruning) por validación cruzada
# ==============================================================================
# Valores de ccp_alpha evaluados
param_grid = {'ccp_alpha':np.linspace(0, 5, 10)}
# Búsqueda por validación cruzada
grid = GridSearchCV(
# El árbol se crece al máximo posible antes de aplicar el pruning
estimator = DecisionTreeClassifier(
max_depth = None,
min_samples_split = 2,
min_samples_leaf = 1,
random_state = 123
),
param_grid = param_grid,
scoring = 'accuracy',
cv = 10,
refit = True,
return_train_score = True
)
grid.fit(X_train_prep, y_train)
fig, ax = plt.subplots(figsize=(8, 3.84))
scores = pd.DataFrame(grid.cv_results_)
scores.plot(x='param_ccp_alpha', y='mean_train_score', yerr='std_train_score', ax=ax)
scores.plot(x='param_ccp_alpha', y='mean_test_score', yerr='std_test_score', ax=ax)
ax.set_title("Error de validacion cruzada vs hiperparámetro ccp_alpha");
# Mejor valor ccp_alpha encontrado
# ==============================================================================
grid.best_params_
{'ccp_alpha': np.float64(0.0)}
Una vez identificado el valor óptimo de ccp_alpha, se reentrena el árbol indicando este valor en sus argumentos. Si en el GridSearchCV() se indica refit=True, este reentrenamiento se hace automáticamente y
el modelo resultante se encuentra almacenado en .best_estimator_.
# Estructura del árbol final
# ==============================================================================
modelo_final = grid.best_estimator_
print(f"Profundidad del árbol: {modelo_final.get_depth()}")
print(f"Número de nodos terminales: {modelo_final.get_n_leaves()}")
Profundidad del árbol: 9 Número de nodos terminales: 49
# Error de test del modelo final
# ==============================================================================
predicciones = modelo_final.predict(X = X_test_prep)
accuracy = accuracy_score(
y_true = y_test,
y_pred = predicciones,
normalize = True
)
print(f"El accuracy de test es: {100 * accuracy} %")
El accuracy de test es: 75.0 %
Gracias al proceso de pruning el porcentaje de acierto ha pasando de 71% a 75%.
Importancia de predictores¶
La importancia de cada predictor en modelo se calcula como la reducción total (normalizada) en el criterio de división, en este caso el índice Gini, que consigue el predictor en las divisiones en las que participa. Si un predictor no ha sido seleccionado en ninguna divisón, no se ha incluido en el modelo y por lo tanto su importancia es 0.
print("Importancia de los predictores en el modelo")
print("-------------------------------------------")
importancia_predictores = pd.DataFrame(
{'predictor': modelo_final.feature_names_in_,
'importancia': modelo_final.feature_importances_}
)
importancia_predictores.sort_values('importancia', ascending=False)
Importancia de los predictores en el modelo -------------------------------------------
| predictor | importancia | |
|---|---|---|
| 11 | Price | 0.267291 |
| 7 | CompPrice | 0.173585 |
| 1 | ShelveLoc_Good | 0.163736 |
| 8 | Income | 0.093583 |
| 9 | Advertising | 0.089938 |
| 12 | Age | 0.055480 |
| 6 | US_Yes | 0.039439 |
| 4 | Urban_Yes | 0.035371 |
| 0 | ShelveLoc_Bad | 0.033437 |
| 10 | Population | 0.024170 |
| 5 | US_No | 0.012651 |
| 13 | Education | 0.011319 |
| 2 | ShelveLoc_Medium | 0.000000 |
| 3 | Urban_No | 0.000000 |
Predicción de probabilidades¶
La mayoría de implementaciones de los modelos basados en árboles, entre ellas la de scikit-learn, permiten predicir probabilidades cuando se trata de problemas de clasificación. Es importante entender cómo se calculan estos valores para interpretarlos y utilizarlos correctamente.
En el ejemplo anterior, al aplicar .predict() se devuelve $1$ (ventas elevadas) o $0$ (ventas bajas) para cada observación de test. Sin embargo, no se dispone de ningún tipo de información sobre la seguridad con la que el modelo realiza esta asignación. Con .predict_proba(), en lugar de una clasificación, se obtiene la probabilidad con la que el modelo considera que cada observación puede pertenecer a cada una de las clases.
# Predicción de probabilidades
# ==============================================================================
predicciones = modelo.predict_proba(X = X_test_prep)
predicciones[:5, :]
array([[0. , 1. ],
[1. , 0. ],
[0.10526316, 0.89473684],
[0.05555556, 0.94444444],
[0.10526316, 0.89473684]])
El resultado de .predict_proba() es un array con una fila por observación y tantas columnas como clases tenga la variable respuesta. El valor de la primera columna se corresponde con la probabilidad, acorde al modelo, de que la observación pertenezca a la clase 0, y así sucesivamente. El valor de probabilidad mostrado para cada predicción se corresponde con la fracción de observaciones de cada classe en el nodo terminal al que ha llegado la observación predicha.
Por defecto, .predict() asigna cada nueva observación a la clase con mayor probabilidad (en caso de empate se asigna de forma aleatoria). Sin embargo, este no tiene por qué ser el comportamiento deseado en todos los casos.
# Clasificación empleando la clase de mayor probabilidad
# ==============================================================================
df_predicciones = pd.DataFrame(data=predicciones, columns=['0', '1'])
df_predicciones['clasificacion_default_0.5'] = np.where(df_predicciones['0'] > df_predicciones['1'], 0, 1)
df_predicciones.head(3)
| 0 | 1 | clasificacion_default_0.5 | |
|---|---|---|---|
| 0 | 0.000000 | 1.000000 | 1 |
| 1 | 1.000000 | 0.000000 | 0 |
| 2 | 0.105263 | 0.894737 | 1 |
Supóngase el siguiente escenario: la campaña de navidad se aproxima y los propietarios de la cadena quieren duplicar el stock de artículos en aquellas tiendas de las que se prevee que tengan ventas elevadas. Como el transporte de este material hasta las tiendas supone un coste elevado, el director quiere limitar esta estrategia únicamente a tiendas para las que se tenga mucha seguridad de que van conseguir muchas ventas.
Si se dispone de las probabilidades, se puede establecer un punto de corte concreto, por ejemplo, considerando únicamente como clase $1$ (ventas altas) aquellas tiendas cuya predicción para esta clase sea superior al 0.8 (80%). De esta forma, la clasificación final se ajusta mejor a las necesidades del caso de uso.
# Clasificación final empleando un threshold de 0.8 para la clase 1.
# ==============================================================================
df_predicciones['clasificacion_custom_0.8'] = np.where(df_predicciones['1'] > 0.8, 1, 0)
df_predicciones.iloc[5:10, :]
| 0 | 1 | clasificacion_default_0.5 | clasificacion_custom_0.8 | |
|---|---|---|---|---|
| 5 | 0.500000 | 0.500000 | 1 | 0 |
| 6 | 0.928571 | 0.071429 | 0 | 0 |
| 7 | 0.055556 | 0.944444 | 1 | 1 |
| 8 | 0.985507 | 0.014493 | 0 | 0 |
| 9 | 0.708333 | 0.291667 | 0 | 0 |
¿Hasta que punto se debe de confiar en estas probabilidades?
Es muy importante tener en cuenta la diferencia entre la visión que tiene el modelo del mundo y el mundo real. Todo lo que sabe un modelo sobre el mundo real es lo que ha podido aprender de los datos de entrenamiento y, por lo tanto, tiene una visión limitada. Por ejemplo, supóngase que, en los datos de entrenamiento, todas las tiendas que están en zona urbana Urban='Yes' tienen ventas altas independientemente del valor que tomen el resto de predictores. Cuando el modelo trate de predecir una nueva observación, si esta está en zona urbana, clasificará a la tienda como ventas elevadas con un 100% de seguridad. Sin embargo, esto no significa que sea inequivocamente cierto, podría haber tiendas en zonas úrbanas que no tienen ventas elevadas pero, al no estar presentes en los datos de entrenamiento, el modelo no contempla esta posibilidad.
Otro ejemplo de como estás probabilidades pueden ser engañosas es el siguiente. Si se deja crecer un árbol hasta que todas las observaciones de entrenamiento están en un nodo terminal distinto, cuando se realice una nueva predicción, independientemente del nodo en el que termine, el 100% de las observaciones (solo hay una) pertenecerán a la misma clase. Como el valor de la probabilidad es el porcentaje de observaciones que pertenecen a cada clase en el nodo terminal, el modelo predicirá siempre con un 100% de probabilidad aunque se equivoque.
Teniendo en cuenta todo esto, hay que considerar las probabilidades generadas por el modelo como la seguridad que tiene este, desde su visión limitada, al realizar las predicciones. Pero no como la probailidad en el mundo real de que así lo sea.
Comparación de árboles frente a modelos lineales¶
La superioridad de los métodos basados en árboles de decisión frente a los métodos lineales depende del problema en cuestión. Cuando la relación entre los predictores y la variable respuesta es aproximadamente lineal, un modelo de tipo regresión lineal funciona bien y supera a los árboles de regresión.
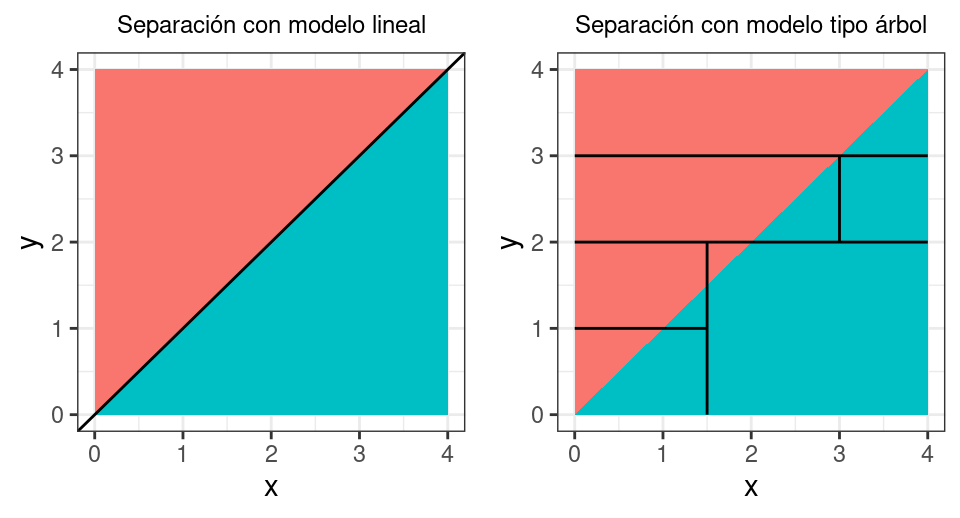
Por contra, si la relación entre los predictores y la variable respuesta es de tipo no lineal y compleja, los métodos basados en árboles suelen superar a las aproximaciones lineales clásicas.

Variables dummy (one-hot-encoding) en modelos de árboles¶
Los modelos basados en árboles de decisión son capaces de utilizar predictores categóricos en su forma natural sin necesidad de convertirlos en variables dummy mediante one-hot-encoding. Sin embargo, en la practica, depende de la implementación que tenga la librería o software utilizado. Esto tiene impacto directo en la estructura de los árboles generados y, en consecuencia, en los resultados predictivos del modelo y en la importancia calculada para los predictores.
El el libro Feature Engineering and Selection A Practical Approach for Predictive Models By Max Kuhn, Kjell Johnson, los autores realizan un experimento en el que se comparan modelos entrenados con y sin one-hot-encoding en 5 sets de datos distintos, los resultados muestran que:
No hay una diferencia clara en términos de capacidad predictiva de los modelos. Solo en el caso de stocastic gradient boosting se parecian ligeras diferencias en contra de aplicar one-hot-encoding.
El entrenamiento de los modelos tarda en promedio 2.5 veces más cuando se aplica one-hot-encoding. Esto es debido al aumento de dimensionalidad al crear las nuevas variables dummy, obligando a que el algoritmo tenga que analizar muchos más puntos de división.
En base a los resultados obtenidos, los autores recomiendan no aplicar one-hot-encoding. Solo si los resultados son buenos, probar entonces si se mejoran al crear las variables dummy.
Otra comparación detallada puede encontrarse en Are categorical variables getting lost in your random forests? By Nick Dingwall, Chris Potts, donde los resultados muestran que:
Al convertir una variable categorica en múltiples variables dummy su importancia queda diluida, dificultando que el modelo pueda aprender de ella y perdiendo así capacidad predictiva. Este efecto es mayor cuantos más niveles tiene la variable original.
Al diluir la importancia de los predictores categóricos, estos tienen menos probabilidad de ser seleccionada por el modelo, lo que desvirtúa las métricas que miden la importancia de los predictores.
Por el momento, en scikit-learn es necesario hacer one-hot-encoding para convertir las variables categóricas en variables dummy.
Extrapolación con modelos de árboles¶
Una límitación importante de los árboles de regresión es que no extrapolan fuera del rango de entrenamiento. Cuando se aplica el modelo a una nueva observación, cuyo valor o valores de los predictores son superiores o inferiores a los observados en el entrenamiento, la predicción siempre es la media del nodo más cercano, independientemente de cuanto se aleje el valor. Vease el siguiente ejemplo en el que se entrenan dos modelos, un modelo lineal y un arbol de regresión, y luego se predicen valores de $X$ fuera del rango de entrenamiento.
import matplotlib.pyplot as plt
import numpy as np
from sklearn.tree import DecisionTreeRegressor
from sklearn.linear_model import LinearRegression
# Datos simulados
# ==============================================================================
X = np.linspace(0, 150, 100)
y = (X + 100) + np.random.normal(loc=0.0, scale=5.0, size=X.shape)
X_train = X[(X>=50) & (X<100)]
y_train = y[(X>=50) & (X<100)]
X_test_inf = X[X < 50]
y_test_inf = y[X < 50]
X_test_sup = X[X >= 100]
y_test_sup = y[X >= 100]
fig, ax = plt.subplots(figsize=(7, 4))
ax.plot(X_train, y_train, c='black', linestyle='dashed', label = "Datos train")
ax.axvspan(50, 100, color='gray', alpha=0.2, lw=0)
ax.plot(X_test_inf, y_test_inf, c='blue', linestyle='dashed', label = "Datos test")
ax.plot(X_test_sup, y_test_sup, c='blue', linestyle='dashed')
ax.set_title("Datos de entrenamiento y test")
plt.legend();
# Modelo lineal
modelo_lineal = LinearRegression()
modelo_lineal.fit(X_train.reshape(-1, 1), y_train)
# Modelo árbol
modelo_tree = DecisionTreeRegressor(max_depth=4)
modelo_tree.fit(X_train.reshape(-1, 1), y_train)
# Predicciones
prediccion_lineal = modelo_lineal.predict(X.reshape(-1, 1))
prediccion_tree = modelo_tree.predict(X.reshape(-1, 1))
fig, ax = plt.subplots(figsize=(7, 4))
ax.plot(X_train, y_train, c='black', linestyle='dashed', label = "Datos train")
ax.axvspan(50, 100, color='gray', alpha=0.2, lw=0)
ax.plot(X_test_inf, y_test_inf, c='blue', linestyle='dashed', label = "Datos test")
ax.plot(X_test_sup, y_test_sup, c='blue', linestyle='dashed')
ax.plot(X, prediccion_lineal, c='firebrick',
label = "Prediccion modelo lineal")
ax.plot(X, prediccion_tree, c='orange',
label = "Prediccion modelo árbol")
ax.set_title("Extrapolación de un modelo lineal y un modelo de árbol")
plt.legend();
La gráfica muestra que, con el modelo de árbol, el valor predicho fuera del rango de entrenamiento, es el mismo independientemente de cuanto se aleje X. Una estrategia que permite que los árboles extrapolen es ajustar un modelo lineal en cada nodo terminal, pero no está disponible en Scikit-learn.
Bibliografía¶
Introduction to Statistical Learning, Gareth James, Daniela Witten, Trevor Hastie and Robert Tibshirani
Applied Predictive Modeling Max Kuhn, Kjell Johnson
T.Hastie, R.Tibshirani, J.Friedman. The Elements of Statistical Learning. Springer, 2009
Bradley Efron and Trevor Hastie. 2016. Computer Age Statistical Inference: Algorithms, Evidence, and Data Science (1st. ed.). Cambridge University Press, USA.
Scikit-learn: Machine Learning in Python, Pedregosa et al., JMLR 12, pp. 2825-2830, 2011.
API design for machine learning software: experiences from the scikit-learn project, Buitinck et al., 2013.
Instrucciones para citar¶
¿Cómo citar este documento?
Si utilizas este documento o alguna parte de él, te agradecemos que lo cites. ¡Muchas gracias!
Árboles de decisión con Python: regresión y clasificación por Joaquín Amat Rodrigo, disponible bajo una licencia Attribution-NonCommercial-ShareAlike 4.0 International (CC BY-NC-SA 4.0 DEED) en https://www.cienciadedatos.net/documentos/py07_arboles_decision_python.html
¿Te ha gustado el artículo? Tu ayuda es importante
Tu contribución me ayudará a seguir generando contenido divulgativo gratuito. ¡Muchísimas gracias! 😊
Este documento creado por Joaquín Amat Rodrigo tiene licencia Attribution-NonCommercial-ShareAlike 4.0 International.
Se permite:
-
Compartir: copiar y redistribuir el material en cualquier medio o formato.
-
Adaptar: remezclar, transformar y crear a partir del material.
Bajo los siguientes términos:
-
Atribución: Debes otorgar el crédito adecuado, proporcionar un enlace a la licencia e indicar si se realizaron cambios. Puedes hacerlo de cualquier manera razonable, pero no de una forma que sugiera que el licenciante te respalda o respalda tu uso.
-
No-Comercial: No puedes utilizar el material para fines comerciales.
-
Compartir-Igual: Si remezclas, transformas o creas a partir del material, debes distribuir tus contribuciones bajo la misma licencia que el original.

